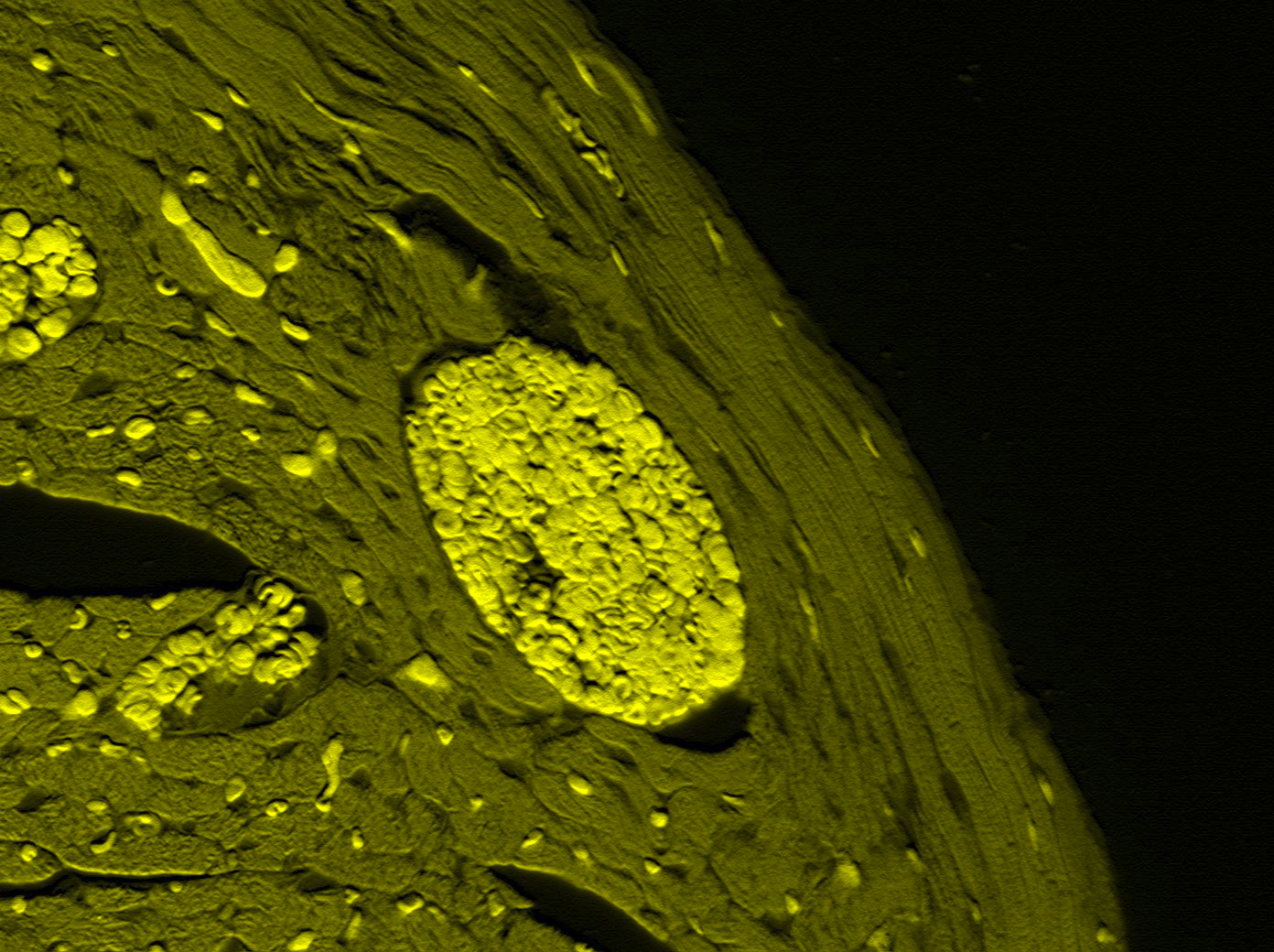
Bioquímica y BMC.
El objetivo final de esta área del BIFI es entender y controlar sistemas biológicos que dependen de proteínas de interés para aplicaciones químicas, biotecnológicas, farmacológicas y biomédicas. El conocimiento del comportamiento de las proteínas a los niveles molecular y celular permite la interpretación de los mecanismos macroscópicos de las funciones celulares en las que están implicados, pero se desconocen todavía muchos de los parámetros que controlan estos procesos. Las proteínas adoptan una estructura tridimensional organizada íntimamente relacionada con su función, que puede ser regulada mediante la interacción con otras biomoléculas y/o pequeñas moléculas orgánicas. Las disposiciones estructurales defectuosas pueden impedir las interacciones de las proteínas con otras moléculas, provocando diversas patologías en los seres humanos.
Muchas otras enfermedades son producidas también por virus y microorganismos infecciosos, y una forma de detenerlos puede ser el bloqueo de algún paso de su ciclo vital que implica a una biomolécula proteica. Además, pequeñas moléculas del medio ambiente pueden funcionar también como inhibidores o activadores tóxicos de actividades particulares en diferentes organismos, y en muchos casos pueden ser usadas como efectores de la expresión de algunos genes y de la producción y acción de determinadas proteínas. Las líneas de investigación en Bioquímica y Biología Molecular y Celular en el BIFI estudian distintos sistemas biológicos implicados en rutas moleculares clave que sirven como modelos de otros sistemas, combinando metodologías clásicas de este área con métodos biofísicos y computacionales. Las aplicaciones del conocimiento obtenido se utilizan adicionalmente para controlar y modular el comportamiento de determinados sistemas con beneficios para la sociedad.
- Regulación génica, fisiología y aplicaciones biotecnológicas de cianobacterias
- Biología evolutiva y genómica de las plantas
- Genómica funcional del sistema OXPHOS (GENOXPHOS)
- Desarrollo de antimicrobianos y mecanismos de resistencia
- Apoptosis y metabolismo
- Genómica computacional y Bio-medicina de sistemas
- Regulación génica, fisiología y aplicaciones biotecnológicas de cianobacterias
- Biología evolutiva y genómica comparada de plantas
- Genómica funcional del sistema OXPHOS (GENOXPHOS)
- Descubrimiento y desarrollo de antimicrobianos y mecanismos de resistencia (D2AMR)
- Apoptosis y metabolismo
- Genómica computacional y Bio-medicina de sistemas
Regulación génica, fisiología y aplicaciones biotecnológicas de cianobacterias
Responsable de la Línea de Investigación:
María F. Fillat Castejón
Investigadores:
Teresa Bes Fustero (Profesora titular)
Emma Sevilla Miguel (Contratado doctor)
Ana Alonso Simón (Ayudante doctor)
Jorge Guío Martínez (Contratado FPU)
Irene Oliván Muro (Contratada FPU)
Inés Federío Zalaya (Contratada programa Investigo)
RESUMEN
Las cianobacterias son microorganismos que realizan la fotosíntesis oxigénica y son capaces de colonizar los hábitats más extremos. Debido a su abundancia y ubicuidad, las cianobacterias juegan un papel fundamental en los ciclos del carbono y del nitrógeno y constituyen la base de la cadena trófica en ecosistemas acuáticos. Algunas especies de cianobacterias son capaces de fijar el nitrógeno atmosférico y se han utilizado en la producción de fertilizantes. Así mismo, el uso de cianobacterias para la producción de biodiesel o en la eliminación de metales pesados de aguas residuales son áreas de trabajo en continuo desarrollo.
La línea de investigación estudia la regulación del metabolismo del hierro en las cianobacterias y su relación con el metabolismo del nitrógeno, el estrés oxidativo, la producción de cianotoxinas y la formación de biofilms. Todos estos procesos están interrelacionados mediante una familia de reguladores transcripcionales denominada FUR (ferric uptake regulator). La mayor parte de las cianobacterias expresan tres parálogos FUR denominados FurA (Fur), Zur (FurB) y PerR (FurC). Aunque la faceta mejor estudiada de estas proteínas es la de regulador transcripcional, en cianobacterias tienen un carácter multifuncional, actuando mediante diversas estrategias no bien caracterizadas, complementando su actividad como reguladores transcripcionales.
Por otra parte, los trabajos del grupo de investigación han demostrado que las proteínas FUR son reguladores globales en cianobacterias, que a su vez controlan a otros reguladores, sistemas de dos componentes y factores sigma. Por ello, se está trabajando en la identificación de nuevas redes reguladoras mediadas por proteínas FUR, caracterizando una serie de reguladores modulados por parálogos FUR cuya función es desconocida.
Otro de los campos de estudio del grupo es la formación de biofilms de cianobacterias, por las implicaciones medioambientales y las aplicaciones que tienen estas formaciones. Para ello se está usando como modelo la cianobacteria filamentosa fijadora de nitrógeno Anabaena sp. PCC7120.
El estudio del potencial de las cianobacterias en biorremediación, en concreto de su capacidad de biodegradar lindano o γ-hexaclorociclohexano (γ-HCH) y sus isómeros (α-,β-,δ-HCH) ha puesto de manifiesto que mutantes de sobreexpresión de FurC en Anabaena PCC7120, tienen una mayor capacidad de biorremediación. La contaminación por hexaclorociclohexano es un problema a nivel mundial, que afecta muy directamente a la Comunidad Autónoma de Aragón debido a la actividad que la industria productora de lindano, Inquinosa, llevó a cabo en Sabiñánigo hace unos años y a que los vertidos que generó siguen almacenados en territorio aragonés. Los estudios realizados por el grupo en los últimos años, han permitido avanzar en la comprensión de la regulación de la expresión de los genes implicados en la degradación del lindano y sus isómeros, e identificar un gen candidato para el diseño de un biosensor. Este biosensor podría permitir una alerta temprana de riesgo en el caso de que estos organoclorados pasaran a cuerpos de agua. En este contexto, se va a trabajar en i) el desarrollo de un prototipo de biosensor de HCH de células enteras, y ii) la identificación de los intermediarios de la ruta de degradación de lindano en cianobacterias.
El grupo de investigación persigue como objetivos principales:
– Identificación de redes reguladoras mediadas por la familia FUR en cianobacterias, y caracterización de nuevos reguladores transcripcionales que forman parte de estas redes y tienen funciones desconocidas.
– Estudio de la formación de biofilms en Anabaena: condiciones que facilitan su formación, genes implicados y potenciales usos de los biofilms en biorremediación y biofertilización.
– Caracterización del regulador Fur del patógeno Clostridium difficile y evaluación de esta proteína como potencial diana terapeúticas mediante la alteración de su actividad mediante el escrutinio de quimiotecas.
-Aplicación de las cianobacterias como organismos relevantes en biorremediación. Concretamente se está estudiando la capacidad de la cianobacteria modelo Anabaena PCC7120 de degradar lindano y sus isómeros. Estos estudios han conducido a la obtención de información que puede utilizarse para desarrollar un biosensor para alerta temprana de presencia de lindano en aguas.
PUBLICACIONES RELEVANTES
1.Unbalancing Zur (FurB)-mediated homeostasis in Anabaena sp. PCC7120: Consequences on metal trafficking, heterocyst development and biofilm formation. Olivan-Muro I, Sarasa-Buisan C, Guio J, Arenas J, Sevilla E, Fillat MF. Environ Microbiol. 2023 Jun 14. doi: 10.1111/1462-2920.16434.
2. Responses of Anabaena sp. PCC7120 to lindane: Physiological effects and differential expression of potential lin genes. Guío J, Fillat MF, Peleato ML, Sevilla E. Microbiology open. 2023 Jun;12(3):e1355. doi: 10.1002/mbo3.1355.
3. Expanding the FurC (PerR) regulon in Anabaena (Nostoc) sp. PCC 7120: Genome-wide identification of novel direct targets uncovers FurC participation in central carbon metabolism regulation. Sarasa-Buisan C, Guío J, Peleato ML, Fillat MF, Sevilla E. PLoS One. 2023 Aug 7;18(8):e0289761. doi: 10.1371/journal.pone.0289761. eCollection 2023.
4. Metal binding and oligomerization properties of FurC (PerR) from Anabaena sp. PCC7120: an additional layer of regulation? Sarasa-Buisan C, Emonot E, Martínez-Júlvez M, Sevilla E, Velázquez-Campoy A, Crouzy S, Bes MT, Michaud-Soret I, Fillat MF. Metallomics. 2022 Oct 20;14(10):mfac077. doi: 10.1093/mtomcs/mfac077.
5. FurC (PerR) from Anabaena sp. PCC7120: a versatile transcriptional regulator engaged in the regulatory network of heterocyst development and nitrogen fixation. Sarasa-Buisan C, Guio J, Broset E, Peleato ML, Fillat MF, Sevilla E. Environ Microbiol. 2022 Feb;24(2):566-582. doi: 10.1111/1462-2920.15552.
6. Thioredoxin Dependent Changes in the Redox States of FurA from Anabaena sp. PCC 7120. Guío J, Bes MT, Balsera M, Calvo-Begueria L, Sevilla E, Peleato ML, Fillat MF. Antioxidants (Basel). 2021 Jun 4;10(6):913. doi: 10.3390/antiox10060913.
7. Fur-like proteins: beyond the Fur uptake regulator (Fur) paralog. Sevilla E, Bes MT, Peleato Ml, Fillat MF. Archives in Biochemistry and Biophysics,701: 108770. doi: 10.1016/j.abb.2021.108770.
8. 2-oxoglutarate modulates the affinity of FurA for the ntcA promoter in Anabaena sp. PCC 7120. Guio, Jorge; Sarasa-Buisan, Cristina; Velazquez-Campoy, Adrian; et ál. FEBS Lett. 2020 Jan;594(2):278-289. doi: 10.1002/1873-3468.13610.
9. Regulation by FurC in Anabaena Links the Oxidative Stress Response to Photosynthetic Metabolism. Sevilla E, Sarasa-Buisan C, González A, Cases R, Kufryk G, Peleato ML, Fillat MF. Plant Cell Physiol. 2019 Aug 1;60(8):1778-1789. doi: 10.1093/pcp/pcz094.2.
10. Redox-Based Transcriptional Regulation in Prokaryotes: Revisiting Model Mechanisms. Sevilla E, Bes MT, González A, Peleato ML, Fillat MF. Antioxid Redox Signal. 2019 May 1;30(13):1651-1696. doi: 10.1089/ars.2017.7442. Epub 2018 Sep 18.
11. Transcriptional regulators: valuable targets for novel antibacterial strategies. González A, Fillat MF, Lanas Á. Future Med Chem. 2018 Mar 1;10(5):541-560. doi: 10.4155/fmc-2017-0181. Epub 2018 Feb 20. Review.
12. Molecular basis for the integration of environmental signals by FurB from Anabaena sp. PCC 7120. Sein-Echaluce VC, Pallarés MC, Lostao A, Yruela I, Velázquez-Campoy A, Luisa Peleato M, Fillat MF. Biochem J. 2018 Jan 5;475(1):151-168. doi: 10.1042/BCJ20170692.
PRINCIPALES PROYECTOS DE INVESTIGACIÓN
1.- E35_23R: Grupo de referencia biología estructural. Gobierno de Aragón. Duración: 01/01/2023 – 31/12/2025. Subvención: 37.743 euros. PI: Marta Martínez-Júlvez y Mª Teresa Bes Fustero. Investigadores: 10.
2.- Redes reguladoras implicadas en la respuesta a estrés y la formación de biofilms en cianobacterias. Identificación de nuevas rutas vinculadas a las proteínas FUR. IP María Francisca Fillat Castejón. Agencia Estatal de Investigación. 01/06/2020 – 31/05/2024. Duración: 4 años
3.- Multifuncionalidad de las proteínas FUR en cianobacterias: mecanismos alternativos de regulación del metabolismo y contribución a la formación de biofilms. MINECO. Duración: 01/01/2017 – 31/12/2019. Subvención: 140.000 euros. PI: María F. Fillat Castejón. Investigadores: 5.
4.- B18 BIOLOGÍA ESTRUCTURAL. Gobierno de Aragón. Duración: 01/01/2014 – 31/12/2016. Subvención: 20.609 euros. PI: María Luisa Peleato Sánchez. Investigadores: 16.
5.- Identificación y aplicaciones sintéticas de nuevos biocatalizadores oxidativos en biotecnología industrial. (BIOXCAT). Ministerio de Ciencia e Innovación. Duración: 01/01/2022-31/12/2023. Subvención: 190.900 euros. PI: Patricia Ferreira y Juan Mangas. Investigadores: 5.
Colaboradores
- Dr. I. Michaud-Soret (Institut de Recherche en Technologie et Science pour le Vivant. CEA, Grenoble, Francia)
- Dr. R. Helm (Virginia Tech, USA)
- Dr. Himadri Pakrasi (Washington St. University, USA)
- Conrad Mullineaux (Queen Mary, University of London, UK)
- Dr. I. Luque (Instituto de Bioquímica vegetal y Fotosíntesis, CSIC, Sevilla, España)
- Dr. Giulia Veronesi (ESFR, Grenoble, Francia)
- Dr. A. Lostao (Instituto de Nanociencia de Aragón, Universidad de Zaragoza, España)
- Dr. A. Lanas (Instituto Aragonés de Ciencias de la Salud, Zaragoza, España)
- Dra. María José Bonete (Universidad de Alicante, Alicante, España)
Contacto
María F. Fillat, e-mail: fillat@unizar.es
X del grupo: @cyanofur
Biología evolutiva y genómica comparada de plantas
Responsable de la Línea de Investigación:
Pilar Catalán Rodríguez
Investigadores:
Pilar Catalán Rodríguez
Ernesto Pérez Collazos
Samira Ben-Menni Schuler
Maria Ángeles Decena Rodríguez
Miguel Campos Cáceres
Alba Sotomayor Alge
Yana Malymon
RESUMEN
El objetivo general de nuestra investigación es establecer sistemas de plantas modelo de referencia (Brachypodium) mediante análisis genómico-funcionales y ecológicos, y la potencial transferencia de los resultados obtenidos con ellas a otras plantas de interés agronómico y biocombustible. Complementariamente a ello, profundizamos en la investigación de gramíneas silvestres en hotspots mundiales, desentrañando sus genomas y su evolución, las interacciones de gramíneas con sus hongos endófitos y con el microbioma del suelo y sus implicaciones co-evolutivas y adaptativas. También estudiamos la caracterización genética y la conservación de plantas endémicas o amenazadas.
Línea 1: Genómica comparada y funcional de plantas modelo
 Los objetivos específicos de nuestros estudios en las plantas modelo Brachypodium buscan obtener genomas completos y pan-genomas anotados de diversas especies clave, y dilucidar, a través de genómica comparada funcional y filogenómica de genomas, transcriptomas y elementos involucrados en la expresión génica (factores de transcripción, remodeladores cromatínicos), los mecanismos reguladores asociados con adaptaciones a estreses abióticos (sequía, nutrientes) y a cambios fenotípicos en especies diploides/alopoliploides y anuales/perennes de Brachypodium. También investigamos el impacto de los transposones en la expresión de la diversidad relacionada con rasgos fenotípicos y funcionales ecológicamente relevantes, y la potencial sobredominancia subgenómica y regulación epigenómica que afecta a la expresión génica diferencial tras la poliploidización, y durante el desarrollo y la adaptación ambiental de estas plantas.
Los objetivos específicos de nuestros estudios en las plantas modelo Brachypodium buscan obtener genomas completos y pan-genomas anotados de diversas especies clave, y dilucidar, a través de genómica comparada funcional y filogenómica de genomas, transcriptomas y elementos involucrados en la expresión génica (factores de transcripción, remodeladores cromatínicos), los mecanismos reguladores asociados con adaptaciones a estreses abióticos (sequía, nutrientes) y a cambios fenotípicos en especies diploides/alopoliploides y anuales/perennes de Brachypodium. También investigamos el impacto de los transposones en la expresión de la diversidad relacionada con rasgos fenotípicos y funcionales ecológicamente relevantes, y la potencial sobredominancia subgenómica y regulación epigenómica que afecta a la expresión génica diferencial tras la poliploidización, y durante el desarrollo y la adaptación ambiental de estas plantas.
Línea 2: Sistemática y evolución de angiospermas
Utilizando los avances genómicos obtenidos en Brachypodium, abordamos el análisis de los genomas apenas explorados de gramíneas pascícolas y forrajeras de alto valor ecológico y económico (género Festuca y subfamilia Pooideae), mediante aproximaciones de genómica comparada y funcional. En este contexto analizamos también las interacciones gramínea-endófito. Para ello, secuenciaremos también genomas de especies fúngicas del género Epichloë, investigando la filogenómica de estos hongos haplo/heteroploides, y su posible co-evolución con sus gramíneas hospedadoras. Complementariamente, investigaremos mediante análisis metabolómicos los beneficios del mutualismo en ecotipos de Festuca cuyos endófitos les proporcionen ventajas adaptativas y agronómicas, incluida la síntesis de alcaloides con función insecticida, para su selección y posible registro como cultivares en la mejora de pastos, céspedes y cubiertas herbáceas.
Línea 3: Genética, ecología y conservación de plantas en Aragón y otras regiones
 Nuestra estrategia investigadora incluye además nuestro compromiso científico y social en estudios de conservación genética (y genómica) de plantas endémicas y amenazadas, tanto de Aragón como de otras regiones. Para ello emplearemos colecciones de herbario, datos de genética (y genómica) poblacional, bio-/filogeografía y modelado de nicho ecológico, así como análisis de biología de la reproducción y técnicas de conservación in-situ y ex-situ, y la divulgación de estos resultados a la sociedad. Nuestras contribuciones constituyen una valiosa herramienta de transferencia de resultados de investigación básica a la investigación aplicada en la conservación de la flora que permite una mejor gestión de las especies en riesgo de extinción y el seguimiento y control de las especies invasoras.
Nuestra estrategia investigadora incluye además nuestro compromiso científico y social en estudios de conservación genética (y genómica) de plantas endémicas y amenazadas, tanto de Aragón como de otras regiones. Para ello emplearemos colecciones de herbario, datos de genética (y genómica) poblacional, bio-/filogeografía y modelado de nicho ecológico, así como análisis de biología de la reproducción y técnicas de conservación in-situ y ex-situ, y la divulgación de estos resultados a la sociedad. Nuestras contribuciones constituyen una valiosa herramienta de transferencia de resultados de investigación básica a la investigación aplicada en la conservación de la flora que permite una mejor gestión de las especies en riesgo de extinción y el seguimiento y control de las especies invasoras.
PUBLICACIONES RELEVANTES
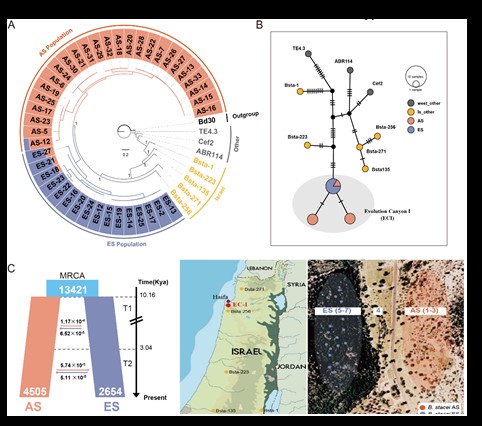
1. Mu W, Li K, Yang Y, Breiman A, Yang J, Wu Y, Wu S, Zhu M, Liu J, Nevo E, Catalán P. 2023. Scattered differentiation of unlinked loci across the genome underlines ecological divergence of the selfing grass Brachypodium stacei. PNAS 120: e2304848120
2. Mu W, Li K, Yang Y, Breiman A, Yang J, Wu J, Zhu M, Wang S, Catalán P, Nevo E, Liu J. 2023. Subgenomic stability of progenitor genomes during repeated allotetraploid origins of the same grass Brachypodium hybridum. Molecular Biology Evolution (accepted).
3. Campos M, Kelley E, Gravendeel B, Médail F, Christenhusz JM, Fay MF, Catalán P, Leitch IJ, Forest F, Wilkin P, Viruel J. 2023. Genomic, spatial and morphometric data for discrimination of four species in the Mediterranean Tamus clade os yams (Dioscorea, Dioscoreaceae). Annals of Botany 131(4):635-654.
4. Scarlett V, Lovell J, Shao M, Phillips J, Shu S, Lusinska J, Goodstein D, Jenkins J, Grimwood J, Barry K, Chalhoub B, Schmutz J, Hasterok R, Catalan P, Vogel J. 2022. Multiple origins, one evolutionary trajectory: gradual evolution characterizes distinct lineages of allotetraploid Brachypodium. Genetics, 10.1093/genetics/iyac146.
5. Sancho R, Catalán P, Contreras-Moreira B, Juenger TE, Des Marais DL. 2022. Patterns of pan-genome occupancy and gene co-expression under water-deficit in Brachypodium distachyon. Molecular Ecology 31:5285-5306. doi: 10.1111/mec.16661
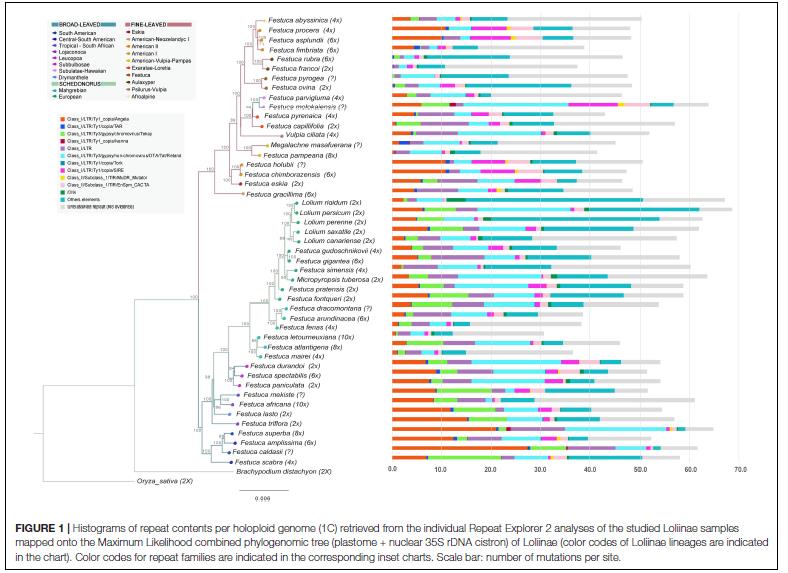
6. Moreno-Aguilar MF, Inda LA, Sánchez A, Arnelas I, Catalán P. 2022. Evolutionary dynamics of the repeatome explains contrasting differences in genome sizes and hybrid and polyploid origins of grass Loliinae lineages. Frontiers in Plant Sciences 13: 901733. doi: 10.3389/fpls.2022.901733
7. Hasterok R, Catalan P, Hazen SP, Roulin AC, Vogel JP, Wang K, Mur LAJ. 2022. Brachypodium: Twenty years as a grass biology model system; the way forward? Trends in Plant Science 27: 1002-1016. https://doi.org/10.1016/j.tplants.2022.04.008.
8. Sancho R, Inda LA, Diaz-Perez A, Des Marais D, Gordon SP, Vogel JP, Lusinska J, Hasterok R, Contreras-Moreira B, Catalán P. 2022. Tracking the ancestry of known and ‘ghost’ homeologous subgenomes in model grass Brachypodium polyploids. The Plant Journal 109(6):1535-1558. doi: 10.1111/tpj.15650. Epub 2022 Feb 8. PMID: 34951515.
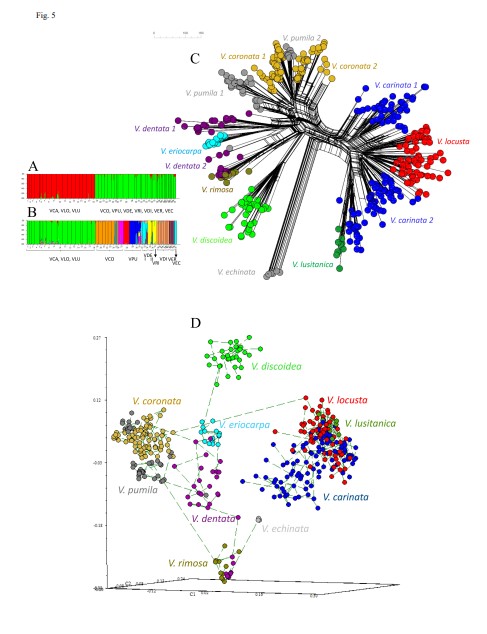
9. Arnelas I, Pérez-Collazos E, López-Martínez J, Devesa JA, Catalán P. 2022. Molecular systematics of Valerianella Mill. (Caprifoliaceae): challenging the taxonomic value of genetically controlled carpological traits. Plants 11: 1276. https://doi.org/10.3390/plants11101276
10. Moreno-Aguilar MF, Inda LA, Sánchez A, Catalán P, Arnelas I. 2022. Phylogenomics and systematics of overlooked Mesoamerican and South American polyploid broad-leaved Festuca grasses differentiate F. sects. Glabricarpae and Ruprechtia and F. subgen. Asperifolia, Erosiflorae, Mallopetalon and Coironhuecu (subgen. nov.). Plants 11, 2303. doi.org/10.3390/plants11172303
PRINCIPALES PROYECTOS DE INVESTIGACIÓN
1. Co-evolution and adaptive speciation grass-endophyte (Festuca, Brachypodium, Epichloë) in a pan-genomic framework (COEVOHOLOGENOME). Ministerio de Ciencia e Innovación. Proyecto PID2022-140074NB-I00. 2023- 2025. IP. P. Catalán. Investigadores participantes del grupo: E. Perez, R. Sancho, M.A. Decena, M. Campos, A. Sotomayor, Y. Malymon. 281.250,00 euros
2. Integrative genomic characterization of the Brachypodium polyploid model to unravel bases of success of polyploidy in flowering plants. Joint Genome Institute. Department of Energy. Government of United States of America. Community Science Program (CSP) proposal 503504. INRA-Evry, University of Silesia, University of Aberystwyth. 2018-2022. IP: P. Catalán. Investigadores participantes: E. Perez, R. Sancho, M.A. Decena, M. Campos, 7,5 TB of genomic and transcriptomic sequencing
3. Gramínea-endófito-microbioma: evolucion, ecología y mejora ecosistémica de pastos y cubiertas herbáceas. Ministerio de Ciencia e Innovación. Proyecto TED2021-131073B-I00. 2022-2024. IP P. Catalán. Investigadores participantes: E. Perez, R. Sancho, M.A. Decena, M. Campos, A. Sotomayor. 113.850,00 euros
4. Gramínea-Microbioma: registro de variedades protegidas de ecotipos de élite y sus microbiomas. Ministerio de Ciencia e Innovación. Proyecto PDC2022-133712-I00. 2022-2024.IP. Catalán. Investigadores participantes: E. Perez, R. Sancho, M. Campos, A. Sotomayor. 149.500,00 euros
COLABORADORES
- John Vogel, Joint Genome Institute (DoE), USA
- David Des Marais, Massachussets Institute of Technology (MIT), USA
- Jianquan Liu, Lanzhou University, China
- Eviatar Nevo, University of Haifa, Israel
- Robert Hasterok, University of Silesia, Polonia
- Jan Hackel, University of Marburg, Alemania
- Itziar Arnelas, Universidad Complutense de Madrid, España
- Iñigo Zabalgogeazcoa, IRNASA-CSIC, España
- Beatriz Rodriguez Vázquez-Aldana, IRNASA-CSIC, España
CONTACTO
Genómica funcional del sistema OXPHOS (GENOXPHOS)
Responsable de la Línea de Investigación:
Patricio Fernández Silva
Pilar Bayona Bafaluy
Investigadores:
Nuria Garrido Pérez
Patricia Meade Huerta
Raquel Moreno Loshuertos
RESUMEN
La actividad de la línea “Genómica funcional del sistema OXPHOS: GENOXPHOS”, adscrita al área de Bioquímica y Biología Molecular, se basa en la comprensión de los factores genéticos y ambientales que controlan la organización y remodelación del sistema OXPHOS mitocondrial. Más concretamente, queremos analizar y entender el efecto de variantes de ensamblaje de supercomplejos (SCs) y de mutaciones en el mtDNA sobre la función OXPHOS y sobre procesos como la capacidad de tumorigénesis o la respuesta a tratamientos con nanopartículas. Así, nuestras principales líneas de trabajo son las siguientes:
- Evaluación de los efectos de alteraciones en el factor de inducción de apoptosis sobre aspectos de la función mitocondrial como la producción de especies reactivas de oxígeno, la organización de complejos y supercomplejos (SCs)respiratorios, la función OXPHOS, la biogénesis mitocondrial o la inducción de muerte celular por la vía de Parthanatos.
- Estudio del papel de la mitocondria en los procesos de tumorigénesis y metástasis, analizando la función mitocondrial y la organización del sistema OXPHOS en líneas tumorales con distinta capacidad invasiva, así como su respuesta al tratamiento con fármacos metabólicos como el dicloroacetato, la metformina o la desoxiglucosa, entre otros.
- Análisis del efecto del incremento de la temperatura sobre el ensamblaje y estabilidad de SCs respiratorios y la función OXPHOS.
- Generación y análisis del funcionamiento de nanotermómetros intracelulares
- Puesta a punto de un sistema de hipertermia magnética local intracelular como terapia antitumoral.
- Estudio molecular de mutaciones en genes codificados en el mtDNA o en el genoma nuclear que afectan al sistema OXPHOS.
- Obtención de células reprogramadas (iPSCs) a partir de fibroblastos para diferenciarlas en los tejidos más comúnmente afectados en pacientes.
PUBLICACIONES RELEVANTES
1.- Moreno-Loshuertos, R., Movilla, N., Marco-Brualla, J., Soler-Agesta, R., Ferreira, P., Enríquez, J. A., & Fernández-Silva, P. A Mutation in Mouse MT-ATP6 Gene Induces Respiration Defects and Opposed Effects on the Cell Tumorigenic Phenotype. International Journal of Molecular Sciences, (2023), 24(2), 1300. https://doi.org/10.3390/ijms24021300
2. Moreno-Loshuertos, R., Marco-Brualla, J., Meade, P., Soler-Agesta, R., Enriquez, J. A., & Fernández-Silva, P. How hot can mitochondria be? Incubation at temperatures above 43 °C induces the degradation of respiratory complexes and supercomplexes in intact cells and isolated mitochondria. Mitochondrion, (2023), 69, 83–94. https://doi.org/10.1016/j.mito.2023.02.002.
3. Ruth Soler-Agesta, Joaquín Marco-Brualla, Patricio Fernández-Silva, Pilar Mozas, Alberto Anel, Raquel Moreno Loshuertos. Transmitochondrial Cybrid Generation Using Cancer Cell Lines. (JoVe) J. Vis. Exp. (2023), (193), e65186, doi:10.3791/65186 (2023). URL: jove.com/video/65186.
4. Y Gu, R. Piñol, R. Moreno-Loshuertos, C. D. S. Brites, J. Zeler, A. Martínez, G. Maurin- Pasturel, P. Fernández-Silva, J. Marco-Brualla, P. Téllez, R. Cases, R. Navarro Belsué, D. Bonvin, L. D. Carlos*, and A. Millán* Local Temperature Increments and Induced Cell Death in Intracellular Magnetic Hyperthermia. ACS Nano 2023, 17, 6822–6832. DOI: 10.1021/acsnano.3c00388.
5. Soler-Agesta R, Marco-Brualla J, Minjárez-Sáenz M, Yim CY, Martínez-Júlvez M, Price MR, Moreno-Loshuertos R, Ames TD, Jimeno J, Anel A. PT-112 Induces Mitochondrial Stress and Immunogenic Cell Death, Targeting Tumor Cells with Mitochondrial Deficiencies. Cancers (Basel). (2022); 14(16), 3851. doi: 10.3390/cancers14163851.
6. Novo N, Romero-Tamayo S, Marcuello C, Boneta S, Blasco-Machin I, Velázquez- Campoy A, Villanueva R, Moreno-Loshuertos R, Lostao A, Medina M, Ferreira P. Beyond a platform protein for the degradosome assembly: The Apoptosis-Inducing Factor as an efficient nuclease involved in chromatinolysis. PNAS Nexus (2022), 2(2), 312. doi: 10.1093/pnasnexus/pgac312.
7. Jiménez-Salvador I, Meade P, Iglesias E, Bayona-Bafaluy P, Ruiz-Pesini E Developmental origins of Parkinson disease: Improving the rodent models..Ageing Res Rev. 2023 Apr;86:101880. doi: 10.1016/j.arr.2023.101880. Epub 2023 Feb 10.
8. Ruiz-Pesini E, Bayona-Bafaluy MP, Sanclemente T, Puzo J, Montoya J, Pacheu-Grau D. Mitochondrial Genetic Background May Impact Statins Side Effects and Atherosclerosis Development in Familial Hypercholesterolemia. Int J Mol Sci. 2022 Dec 28;24(1):471. doi: 10.3390/ijms24010471.
9. Bayona-Bafaluy MP, López-Gallardo E, Emperador S, Pacheu-Grau D, Montoya J, Ruiz-Pesini E. Is population frequency a useful criterion to assign pathogenicity to newly described mitochondrial DNA variants? Orphanet J Rare Dis. 2022 Aug 19;17(1):316. doi: 10.1186/s13023-022-02428-0.
10. Bayona-Bafaluy MP, Montoya J, Ruiz-Pesini E. Oxidative phosphorylation system and cell culture media. Trends Cell Biol. 2021 Aug;31(8):618-620. doi: 10.1016/j.tcb.2021.05.003. Epub 2021 May 26.
PRINCIPALES PROYECTOS DE INVESTIGACIÓN
1.“Nanotermometro intracelular para el estudio termico de la fisiologia celular y terapia del cancer por hipertemia magnetica local:PID2021-124354NB-I00 : Ministerio Ciencia Innovación y Universidades; IPs Angel Millán (CSIC/)/Raquel Moreno Loshuertos (Universidad de Zaragoza). 01/09/2022 al 31/08/2025. 700,00 Euros
2. PID2021-124354NB-I00. Plan Estatal de Investigación Científica, Técnica y de Innovación. Ministerio Ciencia Innovación y Universidades; convocatoria 2021. IPs: Mª Pilar Bayona Bafaluy y Eduardo Ruiz Pesini (Universidad de Zaragoza): 01/09/2022 al 31/08/2025
10.- EUMITOCOMBAT: rational treatment strategies combating mitocondrial oxidative phosphorilation (OXPHOS) disorders. Unión Europea (LSHM-CT-2004-503116). Investigador principal: José Antonio Enríquez.
Colaboradores
- Dr. José Antonio Enriquez. Centro Nacional de Investigaciones Cardiovasculares (CNIC). Madrid-Spain
- Massimo Zeviani. MBU-MRC. Cambridge-UK
- Eva Monleón. Dtpo. de Anatomía e Histología Humanas- UZ
Desarrollo de Antimicrobianos y Mecanismos de Resistencia

Responsable de la Línea de Investigación:
José Antonio Aínsa Claver
Santiago Ramón-García
Investigadores:
José Antonio Aínsa Claver, PI
Santiago Ramón-García, PI
Ainhoa Lucía Quintana, Postdoc
Clara Aguilar Pérez, Doctorando
Ernesto Anoz Carbonell, Doctorando
Ana Cristina Millán Placer, Doctorando
Marta María Gómara Lomero, Doctorando
María Pilar Arenaz Callao, Doctorando
Lara Muñoz Muñoz, Doctorando
Begoña Gracia Díaz, Técnico de Laboratorio
RESUMEN
La línea de investigación Desarrollo de Antimicrobianos y Mecanismos de Resistencia tiene como objetivo estudiar los mecanismos de resistencia a antibióticos de diversos patógenos microbianos y utilizar esta información para identificar nuevas moléculas con actividad antimicrobiana y caracterizar sus mecanismos de acción y resistencia. Este trabajo se financia con fondos públicos conseguidos en convocatorias competitivas nacionales e internacionales.
En los últimos años hemos caracterizado diversas bombas de eflujo de Mycobacterium tuberculosis y hemos contribuido a la identificación de compuestos que evaden el mecanismo de resistencia mediado por las bombas de eflujo y por lo tanto presentan mayor actividad antimicrobiana. Estamos caracterizando nuevas dianas para antimicrobianos, tanto en Mycobacterium como en otros patógenos bacterianos, y explorando nuevas moléculas (péptidos, etc.) como alternativas a los antibióticos convencionales.
Durante los últimos 7 años, hemos publicado 15 artículos, hemos obtenido una patente relacionada con la utilidad diagnóstica de un gen de resistencia, y hemos realizado diversas comunicaciones a congresos nacionales e internacionales.
Nuestra investigación también está incorporando nuevas perspectivas, como lo es la utilización de nanopartículas para administrar antimicrobianos, o las combinaciones de moléculas con actividad antimicrobiana.
PUBLICACIONES RELEVANTES
1.- Discovery of antimicrobial compounds targeting bacterial type FAD synthetases. Sebastián M, Anoz-Carbonell E, Gracia B, Cossio P, Aínsa JA, Lans I, Medina M. J Enzyme Inhib Med Chem. 2018 Dec;33(1):241-254. doi: 10.1080/14756366.2017.1411910. PMID: 29258359
2.- The EU approved antimalarial pyronaridine shows antitubercular activity and synergy with rifampicin, targeting RNA polymerase. Mori G, Orena BS, Franch C, Mitchenall LA, Godbole AA, Rodrigues L, Aguilar-Pérez C, Zemanová J, Huszár S, Forbak M, Lane TR, Sabbah M, Deboosere N, Frita R, Vandeputte A, Hoffmann E, Russo R, Connell N, Veilleux C, Jha RK, Kumar P, Freundlich JS, Brodin P, Aínsa JA, Nagaraja V, Maxwell A, Mikušová K, Pasca MR, Ekins S. Tuberculosis (Edinb). 2018 Sep;112:98-109. doi: 10.1016/j.tube.2018.08.004. PMID: 30205975
3.- Synergy between Circular Bacteriocin AS-48 and Ethambutol against Mycobacterium tuberculosis. Aguilar-Pérez C, Gracia B, Rodrigues L, Vitoria A, Cebrián R, Deboosère N, Song OR, Brodin P, Maqueda M, Aínsa JA. Antimicrob Agents Chemother. 2018 Aug 27;62(9). pii: e00359-18. doi: 10.1128/AAC.00359-18. PMID: 29987141
4.- Boldine-Derived Alkaloids Inhibit the Activity of DNA Topoisomerase I and Growth of Mycobacterium tuberculosis. García MT, Carreño D, Tirado-Vélez JM, Ferrándiz MJ, Rodrigues L, Gracia B, Amblar M, Ainsa JA, de la Campa AG. Front Microbiol. 2018 Jul 24;9:1659. doi: 10.3389/fmicb.2018.01659. PMID: 30087665
5.- Total Synthesis of Ripostatin B and Structure-Activity Relationship Studies on Ripostatin Analogs. Glaus F, Dedić D, Tare P, Nagaraja V, Rodrigues L, Aínsa JA, Kunze J, Schneider G, Hartkoorn RC, Cole ST, Altmann KH. J Org Chem. 2018 Jul 6;83(13):7150-7172. doi: 10.1021/acs.joc.8b00193. PMID: 29542926
6.- New active formulations against M. tuberculosis: Bedaquiline encapsulation in lipid nanoparticles and chitosan nanocapsules. L.De Matteis, D.Jary, A.Lucía, S.García-Embid, I.Serrano-Sevilla, D.Pérez, J.A.Ainsa, F.P.Navarro, J.M. de la Fuente. Chemical Engineering Journal. Volume 340, 15 May 2018, Pages 181-191.
7.- Structure Guided Lead Generation toward Nonchiral M. tuberculosis Thymidylate Kinase Inhibitors. Song L, Merceron R, Gracia B, Quintana AL, Risseeuw MDP, Hulpia F, Cos P, Aínsa JA, Munier-Lehmann H, Savvides SN, Van Calenbergh S. J Med Chem. 2018 Apr 12;61(7):2753-2775. doi: 10.1021/acs.jmedchem.7b01570. PMID: 29510037
8.- Ionophore A23187 shows anti-tuberculosis activity and synergy with tebipenem. Huang W, Briffotaux J, Wang X, Liu L, Hao P, Cimino M, Buchieri MV, Namouchi A, Ainsa JA, Gicquel B. Tuberculosis (Edinb). 2017 Dec;107:111-118. doi: 10.1016/j.tube.2017.09.001. PMID: 29050757
9.- How can nanoparticles contribute to antituberculosis therapy? Costa-Gouveia J, Aínsa JA, Brodin P, Lucía A. Drug Discov Today. 2017 Mar;22(3):600-607. doi: 10.1016/j.drudis.2017.01.011. PMID: 28137645
10.- Antituberculosis drugs: reducing efflux=increasing activity. Rodrigues L, Parish T, Balganesh M, Ainsa JA. Drug Discov Today. 2017 Mar;22(3):592-599. doi: 10.1016/j.drudis.2017.01.002. PMID: 28089787
11.- Structure-Activity Relationships of Spectinamide Antituberculosis Agents: A Dissection of Ribosomal Inhibition and Native Efflux Avoidance Contributions. Liu J, Bruhn DF, Lee RB, Zheng Z, Janusic T, Scherbakov D, Scherman MS, Boshoff HI, Das S, Rakesh, Waidyarachchi SL, Brewer TA, Gracia B, Yang L, Bollinger J, Robertson GT, Meibohm B, Lenaerts AJ, Ainsa J, Böttger EC, Lee RE. ACS Infect Dis. 2017 Jan 13;3(1):72-88. doi: 10.1021/acsinfecdis.6b00158. PMID: 28081607
12.- Identification of Aminopyrimidine-Sulfonamides as Potent Modulators of Wag31-mediated Cell Elongation in Mycobacteria. Vinayak Singh, Neeraj Dhar, János Pató, Gaëlle S. Kolly, Jana Korduláková, Martin Forbak, Joanna C. Evans, Rita Székely, Jan Rybniker, Zuzana Palčeková, Júlia Zemanová, Isabella Santi, François Signorino-Gelo, Liliana Rodrigues, Anthony Vocat, Adrian S. Covarrubias, Monica G. Rengifo, Kai Johnsson, Sherry Mowbray, Joseph Buechler, Vincent Delorme, Priscille Brodin, Graham W. Knott, José A. Aínsa, Digby F. Warner, György Kéri, Katarína Mikušová, John D. McKinney, Stewart T. Cole, Valerie Mizrahi, Ruben C. Hartkoorn. Mol Microbiol. 2017 Jan;103(1):13-25. doi: 10.1111/mmi.13535. PMID: 27677649
13.- Lipid transport in Mycobacterium tuberculosis and its implications in virulence and drug development. Bailo R, Bhatt A, Aínsa JA. Biochem Pharmacol. 2015 Aug 1;96(3):159-67. doi: 10.1016/j.bcp.2015.05.001. PMID: 25986884.
14.- Measuring efflux and permeability in mycobacteria. Rodrigues L, Viveiros M, Aínsa JA. Methods Mol Biol. 2015;1285:227-39. doi: 10.1007/978-1-4939-2450-9_13. PMID: 25779319.
15.- Spectinamides: a new class of semisynthetic antituberculosis agents that overcome native drug efflux. Lee RE, Hurdle JG, Liu J, Bruhn DF, Matt T, Scherman MS, Vaddady PK, Zheng Z, Qi J, Akbergenov R, Das S, Madhura DB, Rathi C, Trivedi A, Villellas C, Lee RB, Rakesh, Waidyarachchi SL, Sun D, McNeil MR, Ainsa JA, Boshoff HI, Gonzalez-Juarrero M, Meibohm B, Böttger EC, Lenaerts AJ. Nat Med. 2014 Feb;20(2):152-8. doi: 10.1038/nm.3458. PMID: 24464186.
PRINCIPALES PROYECTOS DE INVESTIGACIÓN
1.- El fenotipo silente de Mycobacterium tuberculosis: persistencia y latencia. Ministerio de Economía y Competitividad. Universidad de Zaragoza. SAF2017-84839-C2-1-R. 01/01/2018 – 31/12/2020. IP José Antonio Aínsa Claver.
2.- Identification of novel therapies for difficult to treat cystic fibrosis pulmonary infections caused by mycobacteria using an innovative technology: synergy screens of clinically approved drugs. Unión Europea. Universidad de Zaragoza. 01/04/2018 – 31/03/2019. IP: Santiago Ramon Garcia.
3.- NAREB – Nanotherapeutics for antibiotic resistant emerging bacterial pathogens. Unión Europea. Universidad de Zaragoza. 01/02/2014 – 31/07/2018. IP: José Antonio Aínsa Claver.
4.- SAF-2013-48971-C2-2-R: Aplicaciones biomédicas de AS-48: una proteína con amplio espectro de actividad antimicrobiana. MINECO – Ministerio de Economia y Competitividad. Universidad de Zaragoza. 01/01/2014 – 31/07/2018. IP: José Antonio Aínsa Claver.
5.- MM4TB – More medicines for tuberculosis. Unión Europea. Universidad de Zaragoza. 01/02/2011 – 31/01/2016. IP: José Antonio Aínsa Claver.
Colaboradores
- Adela G. De la Campa, Centro Nacional de Microbiología, Instituto de Salud Carlos III (Majadahonda, Madrid, Spain). Desarrollo de inhibidores frente a la topoisomerasa de tuberculosis.
- Mercedes Maqueda, Universidad de Granada (Granada, Spain). Estudio de actividad antimicrobiana de la bacteriocina AS-48.
- Serge van Calenbergh, Universidad de Gent (Gent, Belgium). Nuevos inhibidores de timidilato kinasa de tuberculosis.
- Concepción González-Bello (Universidad de Santiago de Compostela, Santiago de Compostela, Spain). Inhibidores de dehidroquinasas en tuberculosis.
Apoptosis y metabolismo
Responsable de la Línea de Investigación:
José Alberto Carrodeguas Villar
Investigadores:
Beatriz Sáenz de Buruaga/Est. predoc
Laura Bueno Martínez/Est. TFG
Sara García Gadea/Est. TFG
RESUMEN
Nuestro trabajo se ha centrado, durante los últimos años, en varios campos, que se indican a continuación:
1.- Estudio de la función de PSAP/Mtch1 (Presenilin 1-associated protein/mitochondrial carrier homolog 1). PSAP interacciona con la presenilina 1, la cual forma parte del complejo gamma secretasa, implicado en la enfermedad de Alzheimer. PSAP también se conoce como mitochondrial carrier homolog 1 (Mtch1), porque contiene un dominio proteico conservado en transportadores de la membrana interna mitocondrial, aunque se localiza en la membrana externa. Mtch1 induce muerte celular cuando se sobreexpresa en células en cultivo.
Hemos identificado dos isoformas proapoptóticas generadas por splicing alternativo, que se dirigen a la membrana externa mitocondrial mediante varias señales de localización internas y que contienen dos dominios proapoptóticos. Mtch1 puede inducir apoptosis en ausencia de Bax y Bak, miembros proapoptóticos de la familia de Bcl-2. No parece que tenga una función transportadora, pero podría funcionar como un receptor de ligandos todavía desconocidos en la superficie de la mitocondria.
Hemos analizado el knockout de Mtch1 en Drosophila y estamos preparando su publicación, en colaboración con investigadores del Instituto de Investigaciones Biomédicas, CSIC-UAM, en Madrid.
2.- Proteínas Bcl-2. Las proteínas de la familia de Bcl-2 son esenciales en el inicio de la muerte celular, integrando varias señales celulares. Sus funciones principales conocidas dependen de interacciones proteína-proteína para la inducción o la inhibición de los pasos iniciales de la muerte celular. Sin embargo cada vez hay más evidencias de que pueden tener funciones alternativas relacionadas con la regulación del metabolismo. En esta línea, hemos descrito la implicación del dominio transmembrana de Bcl-XL en la dimerización de esta proteína.
3.- PEPCK. Junto con el Dr. Pascual López Buesa, hemos estudiado varios polimorfismos genéticos que afectan a la calidad de la carne de cerdo, habiéndonos centrado en los últimos años en ambas isoformas de la fosfoenolpiruvato carboxikinasa, la citosólica y la mitocondrial. Este trabajo ha evolucionado para estudiar la regulación post-traduccional de esta enzima, esencial para la gluconeogénesis e implicada en patologías como la diabetes o el cáncer. En un trabajo reciente, en colaboración con el Dr. John Denu, del Wisconsin Institute for Discovery, de la Universidad de Wisconsin-Madison y con investigadores de Australia, La Rioja y Harvard, hemos descubierto nuevos mecanismos de regulación posttraduccional de la PEPCK de mamíferos. Este trabajo ha sido publicado recientemente en la revista Molecular Cell.
Estamos concluyendo otra serie de experimentos en esta misma línea.
4.- Enfermedad de Parkinson. Junto con la Dra. Nunilo Cremades. del BIFI, estamos comenzando un proyecto de investigación sobre Parkinson que utiliza un abordaje multidisciplinar, biofísico (Dr. Cremades) y celular (Dr. Carrodeguas), para desarrollar nuevos modelos celulares y utilizar técnicas biofísicas de última generación con el fin de determinar los mecanismos iniciales que producen la agregación de la proteína a-sinucleína en esta patología.
5.- Células madre. Hemos trabajado también en diferenciación y muerte de células madre y vamos a aplicar estos conocimientos en el desarrollo de modelos para Parkinson.
Colaboramos también con otros investigadores del BIFI en distintas líneas de investigación.
PUBLICACIONES RELEVANTES
- Latorre-Muro P, Baeza J, Armstrong, EA, Hurtado-Guerrero R, Corzana F, Wus LE, Sinclair DA, López-Buesa P, Carrodeguas JA, Denu JM (2018). Dynamic acetylation of cytosolic phosphoenolpyruvate carboxykinase toggles enzyme activity between gluconeogenic and anaplerotic reactions. Mol. Cell. 71: 718-732. doi: 10.1016/j.molcel.2018.07.031.
- Latorre P, Varona L, Burgos C, Carrodeguas JA, López-Buesa P. (2017). O-GlcNAcylation mediates the control of cytosolic phosphoenolpyruvate carboxykinase activity via Pgc1α. PLoS One 12: e0179988. doi: 10.1371/journal.pone.0179988.
- Hidalgo J, Latorre P, Carrodeguas JA, Velázquez-Campoy A, Sancho J, López-Buesa P. (2016). Inhibition of Pig Phosphoenolpyruvate Carboxykinase Isoenzymes by 3-Mercaptopicolinic Acid and Novel Inhibitors. PLoS One. 11: e0159002. doi: 10.1371/journal.pone.0159002.
- Escós M, Latorre P, Hidalgo J, Hurtado-Guerrero R, Carrodeguas JA, López-Buesa P. (2016). Kinetic and functional properties of human mitochondrial phosphoenolpyruvate carboxykinase. Biochem Biophys Rep. 7: 124-129. doi: 10.1016/j.bbrep.2016.06.007. Co-corresponding author.
- Latorre P, Burgos C, Hidalgo J, Varona L, Carrodeguas JA, López-Buesa P. (2016). c.A2456C-substitution in Pck1 changes the enzyme kinetic and functional properties modifying fat distribution in pigs. Sci Rep. 6: 19617. doi: 10.1038/srep19617. Co-corresponding author.
- Nelo-Bazán MA, Latorre P, Bolado-Carrancio A, Pérez-Campo FM, Echenique-Robba P, Rodríguez-Rey JC, Carrodeguas JA. (2015). Early growth response 1 (EGR-1) is a transcriptional regulator of mitochondrial carrier homolog 1 (MTCH 1)/presenilin 1-associated protein (PSAP). Gene 578:52-62. doi: 10.1016/j.gene.2015.12.014.
- Echenique-Robba P, Nelo-Bazán MA, Carrodeguas JA. (2013). Reducing the standard deviation in multiple-assay experiments where the variation matters but the absolute value does not. PLoS One 8: e78205. doi: 10.1371/journal.pone.0078205.
- Ospina A, Lagunas-Martínez A, Pardo J, Carrodeguas JA (2011). Protein oligomerization mediated by the transmembrane carboxyl terminal domain of Bcl-XL. FEBS Lett. 585: 2935-42. doi: 10.1016/j.febslet.2011.08.012.
- Conesa C, Doss MX, Antzelevitch C, Sachinidis A, Sancho J, Carrodeguas JA (2012). Identification of specific pluripotent stem cell death–inducing small molecules by chemical screening. Stem. Cell Rev. 2012 Mar;8(1):116-27. doi: 10.1007/s12015-011-9248-4.
- Lamarca V, Marzo I, Sanz-Clemente A, Carrodeguas JA (2008). Exposure of any of two proapoptotic domains of presenilin 1-associated protein/mitochondrial carrier homolog 1 on the surface of mitochondria is sufficient for induction of apoptosis in a Bax/Bak-independent manner. Eur. J. Cell. Biol. 87: 325-34. doi: 10.1016/j.ejcb.2008.02.004.
PRINCIPALES PROYECTOS DE INVESTIGACIÓN
1.- Modulación de las características del músculo esquelético por la fosfoenolpiruvato carboxiquinasa. Facultad de Veterinaria – Universidad de Zaragoza. Pascual López Buesa y José Alberto Carrodeguas Villar. CICYT. 2016-1018.
2.- Modulación de las características del músculo esquelético por la fosfoenolpiruvato carboxiquinasa. Instituto Universitario De Investigación De Biocomputación y Física De Sistemas Complejos – Universidad de Zaragoza. José Alberto Carrodeguas Villar. VIC. INV. – APOYO INV. 2016.
3.- PEPCK y sus efectos sobre el metabolismo, los caracteres productivos y la calidad de la carne y la canal del ganado porcino. Facultad De Veterinaria – Universidad de Zaragoza. Pascual Luis López Buesa. VIC. INV. – APOYO INV. 2015.
4.- Identificación de moléculas bioactivas en células troncales mediante cribado funcional de quimiotecas: herramientas para terapias seguras. Instituto de Biocomputación y Física de Sistemas Complejos. José Alberto Carrodeguas Villar. Universidad de Zaragoza/Ibercaja. 2012-2013.
5.- Proteínas Mtch: regulación transcripcional en humanos y efectos fenotípicos del mutante en Drosophila. Instituto Universitario De Investigación De Biocomputación y Física De Sistemas Complejos. José Alberto Carrodeguas Villar. Universidad de Zaragoza. 2011.
6.- Genética química para la identificación de compuestos bioactivos que promueven diferenciación específica, proliferación o apoptosis en células madre. Instituto de Biocomputación y Física de Sistemas Complejos. Departamento de Bioquímica y Biología Molecular y Celular. Facultad de Ciencias. Universidad de Zaragoza. José Alberto Carrodeguas Villar. Instituto Aragonés de Ciencias de la Salud. 2011.
7.- Regulación de la actividad de proteínas proapoptóticas mitocondriales. Instituto de Biocomputación y Física de Sistemas Complejos. Departamento de Bioquímica y Biología Molecular y Celular. Facultad de Ciencias. Universidad de Zaragoza. José Alberto Carrodeguas Villar. Ministerio de Ciencia e Innovación. 2010.
8.- Identificación de compuestos químicos que inducen diferenciación celular específica o muerte celular apoptótica en células madre embrionarias de ratón (continuación). Instituto de Biocomputación y Física de Sistemas Complejos. Departamento de Bioquímica y Biología Molecular y Celular. Facultad de Ciencias. José Alberto Carrodeguas Villar. Instituto Aragonés de Ciencias de la Salud. 2009-2010.
9.- Identificación de compuestos químicos que inducen diferenciación celular específica o muerte celular apoptótica en células madre embrionarias de ratón (continuación). Instituto de Biocomputación y Física de Sistemas Complejos. Departamento de Bioquímica y Biología Molecular y Celular. Facultad de Ciencias. José Alberto Carrodeguas Villar. Instituto Aragonés de Ciencias de la Salud. 2008-2010.
10.- Mecanismos moleculares de proteínas de la membrana externa mitocondrial similares a transportadores implicadas en apoptosis. Papel en enfermedades degenerativas y en cáncer. Facultad de Veterinaria. Universidad de Zaragoza. José Alberto Carrodeguas Villar. MEC. 2006-2009.
Colaboradores
- Miguel Fernández Moreno and Juan José Arredondo. Instituto de Investigaciones Biomédicas. CSIC-UAM. Madrid.
- Javier Sancho, Milagros Medina, Adrián Velázquez Campoy, Ramón Hurtado-Guerrero, Patricio Fernández Silva, Raquel Moreno Loshuertos. Instituto de Biocomputación y Física de Sistemas Complejos. Departamento de Bioquímica y Biología Molecular y Celular. Facultad de Ciencias. Universidad de Zaragoza.
- José Carlos Rodríguez-Rey. Department of Molecular Biology, University of Cantabria, IDIVAL, Santander, Cantabria, Spain.
- Flor Pérez Campo. Department of Internal Medicine, Hospital U. Marqués de Valdecilla-IDIVAL University of Cantabria, 39008 Santander, Cantabria, Spain.
- John M. . Wisconsin Institute for Discovery, Morgridge Institute for Research, and the Department of 12 Biomolecular Chemistry, University of Wisconsin School of Medicine and Public Health, USA.
- Francisco Corzana. Departamento de Química, Centro de Investigación en Síntesis Química, Universidad de La Rioja.
Genómica computacional y Bio-medicina de sistemas
Responsable de la Línea de Investigación:
Joaquín Sanz
Investigadores:
PhD students:
Mario Tovar
Jorge Cárdenas
Master students:
Ignacio Marchante
Undergrad students:
Santiago Royo
Pablo Pérez
Pilar Cobos.
Alumni:
Jessica Moreira Batista Da Silva
Regina Santesteban Azanza
RESUMEN
En nuestro grupo utilizamos modelos matemáticos para describir enfermedades infecciosas a diferentes escalas de complejidad: desde células y genes hasta individuos y poblaciones. Nuestro principal objetivo es identificar los factores causales, tanto genéticos como ambientales, que configuran la variación en las respuestas inmunitarias a patógenos, así como caracterizar los factores que definen el carácter patológico o funcional de dichas respuestas y estudiar cómo se relacionan con observaciones epidemiológicas.
Para ello, nuestra principal materia prima son datos de secuenciación genómica. Eso incluye datos de variación genética en grandes cohortes humanas, así como transcriptomas y epigenomas de hospedadores y patógenos, tanto en modelos humanos como animales, a menudo a resolución de célula única. En el aspecto epidemiológico, desarrollamos modelos de transmisión de enfermedades comunicables, para interpretar datos tales como registros de incidencia transnacionales, encuestas de prevalencia y resultados de ensayos clínicos. Para lograr estos objetivos, nuestros métodos integran herramientas de genómica computacional, biomedicina de sistemas, bioestadística, ciencia de datos y redes, inferencia bayesiana, epidemiología matemática y física de sistemas complejos.
En la actualidad, hay tres líneas principales de investigación abiertas en el grupo. Estas incluyen la Biología de Sistemas de las interacciones huésped-patógeno en tuberculosis; el desarrollo de herramientas bioinformáticas para el análisis de datos -ómicos a resolución single-cell y el desarrollo de aplicaciones computacionales para el estudio de la genómica del sistema inmunológico.
Biologia de sistemas de tuberculosis
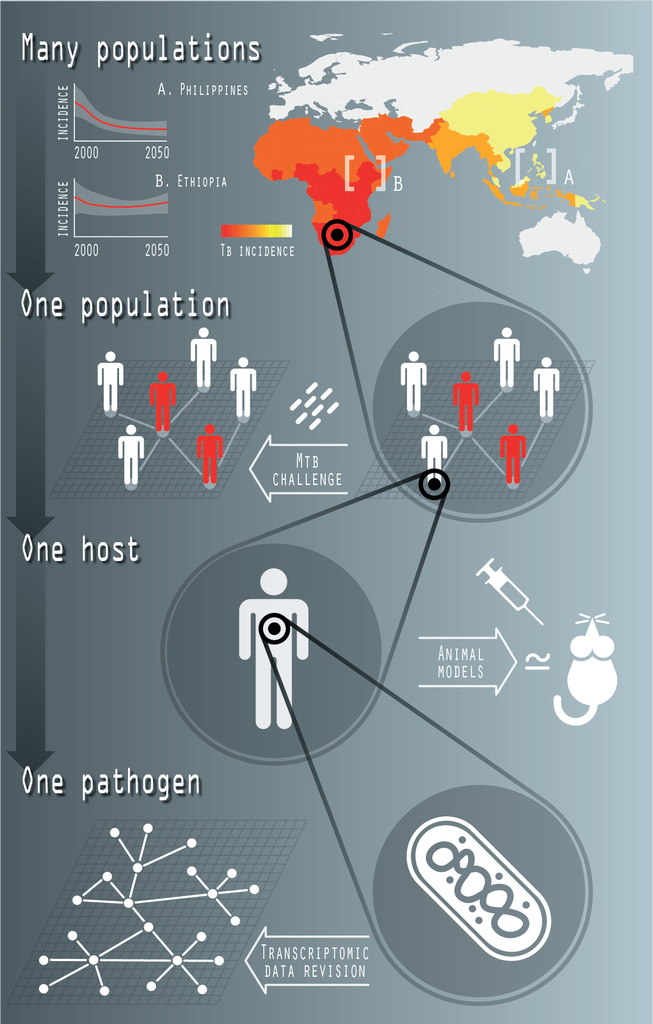 La tuberculosis (TB) es una de las enfermedades infecciosas humanas más antiguas y, con un número estimado de 1,4 millones de muertes en 2019, todavía hoy una de las más mortíferas. Su agente causal, el bacilo Mycobacterium tuberculosis, es posiblemente el más exitoso entre todos los patógenos humanos, considerando su sorprendente capacidad para coexistir con su huésped (se estima que alrededor del 24% de los humanos contemporáneos están infectados con M.tb.) sin comprometer su salud. Entre todas las posibles intervenciones epidemiológicas que se están considerando en la lucha contra la tuberculosis, la introducción de una nueva vacuna capaz de complementar o superar a la vacuna actual del Bacillus Calmette-Guerin (BCG), promete un mayor impacto contra la enfermedad.
La tuberculosis (TB) es una de las enfermedades infecciosas humanas más antiguas y, con un número estimado de 1,4 millones de muertes en 2019, todavía hoy una de las más mortíferas. Su agente causal, el bacilo Mycobacterium tuberculosis, es posiblemente el más exitoso entre todos los patógenos humanos, considerando su sorprendente capacidad para coexistir con su huésped (se estima que alrededor del 24% de los humanos contemporáneos están infectados con M.tb.) sin comprometer su salud. Entre todas las posibles intervenciones epidemiológicas que se están considerando en la lucha contra la tuberculosis, la introducción de una nueva vacuna capaz de complementar o superar a la vacuna actual del Bacillus Calmette-Guerin (BCG), promete un mayor impacto contra la enfermedad.
En nuestro grupo, combinamos enfoques de biología de sistemas, bioinformática y epidemiología matemática para modelar diferentes aspectos asociados a la infección por M.tb. que operan a diferentes niveles de complejidad. Algunos ejemplos incluyen desde la caracterización genómica de la memoria inmune innata inducida por micobacterias hasta el modelado epidemiológico de la transmisión de la TB a nivel transnacional, para interpretar los resultados de ensayos clínicos y evaluar el impacto de intervenciones epidemiológicas tales como la introducción de nuevas y mejores vacunas.
Fig. 1: Biología de sistemas de la TB. La actividad reciente del grupo en este tema incluye la caracterización de redes reguladoras transcripcionales, a nivel de bacteria individual (un patógeno); el estudio de la inmunidad entrenada innata en colaboración con M. Divangahi (McGill) y su equipo (un huésped); la caracterización de la arquitectura genética de la respuesta de los macrófagos humanos a M.tb. (una población), y el desarrollo de modelos matemáticos de transmisión de la TB, a escala supranacional (muchas poblaciones).
Single-cell -omics: modelización y análisis de datos.
En 2009, Fuchou Tang y sus colaboradores fueron los primeros en utilizar RNA-seq a resolución single-cell, de un blastómero de ratón. Desde entonces, el campo ha experimentado un progreso espectacular en técnicas de microfluidos, automatización de la preparación de librerías y multiplexación, hasta el punto que ahora es posible, y relativamente asequible, compilar datasets que contienen los transcriptomas de cientos de miles de células. Estas mejoras experimentales abren la posibilidad de abordar problemas biológicos más profundos mediante el uso de diseños experimentales complejos.
En el grupo combinamos ciencia de datos, modelado estadístico y métodos de redes complejas para proponer pipelines mejorados para el análisis de datos transcriptómicos a resolución single-cell, prestando especial atención al desarrollo de aplicaciones para la caracterización de respuestas a infección, entrenamiento inmunológico, y vacunación en células inmunes y sus precursores hematopoyéticos.
Genómica computacional del sistema inmunitario
Las respuestas inmunitarias a las agresiones externas son complejas y variables entre individuos. La idoneidad de la respuesta inmune a una infección a menudo depende de su intensidad, rapidez y especificidad; y todas estas son características dinámicas que emergen de una interacción compleja entre numerosos factores causales distintos. Estos factores provienen a su vez de la genética del huésped y del patógeno, así como del contexto ambiental en el que se produce su contacto.
En el laboratorio estudiamos los vínculos de causalidad que conectan los genotipos y las variables ambientales con las respuestas inmunitarias a la infección. Nuestro principal objetivo es la identificación de los componentes de tales respuestas que se ven más fuertemente afectados por dichos factores causales, así como sus implicaciones evolutivas y clínicas.
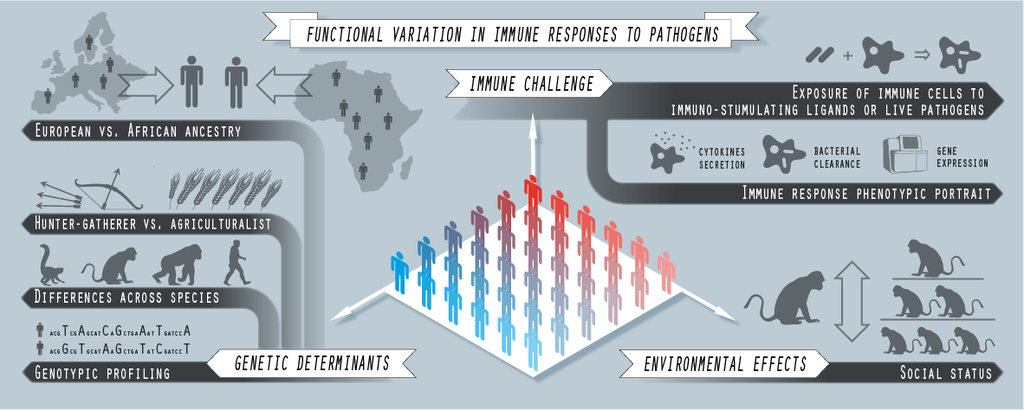 Fig. 2. Genómica computacional del Sistema inmunitario. Esquema gráfico de los enfoques experimentales implementados en algunos de los últimos proyectos en los que hemos trabajado recientemente, en colaboración con L. Barreiro (U. Chicago), J. Tung (Duke), y sus equipos
Fig. 2. Genómica computacional del Sistema inmunitario. Esquema gráfico de los enfoques experimentales implementados en algunos de los últimos proyectos en los que hemos trabajado recientemente, en colaboración con L. Barreiro (U. Chicago), J. Tung (Duke), y sus equipos
Publicaciones relevantes
- Primate innate immune responses to bacterial and viral pathogens reveals an evolutionary trade-off between strength and specificity. Hawash, M., Sanz, J, Grenier, J. C., Kohn, J., Yotova, V., Johnson, Z., … & Barreiro, L. B. (2020). Proceedings of the National Academy of Sciences, 2021.
- tuberculosis Reprograms Hematopoietic Stem Cells to Limit Myelopoiesis and Impair Trained Immunity. Khan1, Downey, Sanz, J., Kaufmann, Blankenhaus, Pacis, … & Divangahi (2020). Cell, 183(3), 752-770.
- Social history and exposure to pathogen signals modulate social status effects on gene regulation in rhesus macaques. Sanz, J., Maurizio, P. L., Snyder-Mackler, N., Simons, N. D., Voyles, T., Kohn, J., … & Barreiro, L. B. (2020). Proceedings of the National Academy of Sciences, 117(38), 23317-23322.
- Bridging the gap between efficacy trials and model-based impact evaluation for new tuberculosis vaccines. Tovar, M., Arregui, S., Marinova, D., Martín, C., Sanz, J., & Moreno, Y. Nature Communications, 10(1), 1-10. (1 co-last author) (2019).
- Natural selection contributed to immunological differences between hunter-gatherers and agriculturalists. Harrison, G.F., Sanz, J., Boulais, J., Mina, M.J., Grenier, J.C., Leng, Y., Dumaine, A., Yotova, V., Bergey, C. M., Nsobya, S.L., Elledge, S.J., Schurr, E., Quintana-Murci, L., Perry, G.H., Barreiro, L.B. Nature Ecology and Evolution 1253-1264 3(8) (2019).
- Spotting the old foe—revisiting the case definition for TB. Houben, R.M., Esmail, H., Emery, J.C., Joslyn, L.R., McQuaid, C.F., Menzies, N.A., Sanz, J., Shrestha, S., White, R.G., Yang, C. and Cobelens, F., 2019. The Lancet Respiratory Medicine, 7(3), pp.199-201.
- Social status alters chromatin accessibility and the gene regulatory response to glucocorticoid stimulation in rhesus macaques. Snyder-Mackler, N., Sanz, J., Kohn, J. N., Voyles, T., Pique-Regi, R., Wilson, M. E., … & Tung, J. (2019). Proceedings of the National Academy of Sciences, 116(4), 1219-1228.
- Genetic and evolutionary determinants of human population variation in immune responses. Sanz, J., Randolph, H. E., & Barreiro, L. B. (2018). Current opinion in genetics & development, 53, 28-35.
- Data-driven model for the assessment of Mycobacterium tuberculosis transmission in evolving demographic structures. Arregui, S., Iglesias, M. J., Samper, S., Marinova, D., Martin, C., Sanz, J. , & Moreno, Y. 1 . (1 co-last author). (2018). Proceedings of the National Academy of Sciences, 115(14), E3238-E3245.
- BCG educates hematopoietic stem cells to generate protective innate immunity against tuberculosis. Kaufmann, E. 1, Sanz, J. 1, Dunn, J. L. 1, Khan, N., Mendonça, L. E., Pacis, A., … & Mailhot-Léonard, F. (1 co-first author). (2018). Cell, 172(1-2), 176-190.
- Social status alters immune regulation and response to infection in macaques. N. Snyder-Mackler1, J. Sanz1, J.N. Kohn, J.F. Brinkworth, S. Morrow, A.O. Shaver, J.C. Grenier, R. Pique-Regi, Z.P. Johnson, M.E. Wilson, L.B. Barreiro2 & J. Tung2 (1 co-first author; 2 co-last author); Science, 354 (6315), 1041-1045.
- Genetic ancestry and natural selection drive population differences in immune responses to pathogens. Y. Nédéléc1, J. Sanz1, G. Baharian1, Z.A. Szpiech, A. Pacis, A. Dumaine, J.C. Grenier, A. Freiman, J. Sams, S. Hebert, A. Pagé-Sabourin, F. Luca, R. Blekhman, R.D. Hernández, R. Piqué-Regi, J. Tung, V. Yotova & L.B.B. Barreiro, (1 co-first author). Cell 167-3, p657–669.e21 (2016) (The paper was selected for the Issue cover).
Principales proyectos
- PID2019-106859GA-I00: Enfoques sistémicos a los mecanismos de defensa del hospedador ante enfermedad e infección en M. tuberculosis: causas genéticas y evaluación de impacto en nuevas vacunas, 2020-2023 Spanish Ministry of Science and Innovation (MICINN), Principal Investigator: Joaquín Sanz.
- Bio-computational approaches applied to the development of TB vaccines: epidemiological modeling, efficacy simulations and immunogenetics analyses. Government of Aragon, Spain. Grant LMP117-18. 2019-2020. PI: Yamir Moreno.
- Multi-scale approaches to Tuberculosis infection: mathematical epidemiology and functional genomics. National Programme for Recruitment and Incorporation of Human Resources 2018, subprogramme “Ramón y Cajal”. RYC-2017-23560. 2019-2024. Principal Investigator: Joaquín Sanz.
- Stress and the Genome: Testing the Impact of Social Effects on Gene Regulation. National Institute of Health NIH (USA) Project # 1R01GM102562-01. (2012-2022). Principal Investigator: Dr. Jenny Tung. (Duke University)
Colaboradores
- Maziar Divangahi, McGill University, Canada.
- Eva Kaufmann, McGill University, Canada.
- Luis Barreiro, University of Chicago, USA.
- Genelle Harrison, University of Colorado, USA.
- Jenny Tung, Duke University, Durham, USA.
- Bana Jabri, University of Chicago, USA.
- Valentina Discepolo, Universitá Federico II, Naples, Italy.
- Carlos Martín, unizar.
- Nacho Aguiló, unizar.
- Jesús Gonzalo-Asensio, unizar.
- Yamir Moreno, unizar.
- Pierpaolo Bruscolini, unizar.
- Mario Floría, unizar.
- Jesús Gómez-Gardeñes, unizar.
- Sandro Meloni, IFISC, Palma de Mallorca.
Contacto
Jsanz@bifi.es
https://sanzlab.wordpress.com/