Biofísica.

Los investigadores en el área de Biofísica en el BIFI usan técnicas experimentales y computacionales en un ambiente interdisciplinario para comprender el comportamiento de los sistemas biológicos. El estudio en esta área abarca desde moléculas (proteínas, ácidos nucleicos, moléculas pequeñas, etc.) a los organismos y ecosistemas enteros, desde un punto de vista cuantitativo. Las proteínas y los organismos con relevancia biotecnológica y/o biomédica son de especial interés. La investigación en esta área tiene muchas aplicaciones en diferentes campos:
Biotecnología (estabilización de proteínas y el modelado de las mismas, diseño de la función de proteínas, máquinas moleculares, nanoestructuras, modelado molecular, transporte electrónico, catálisis, la interacción electromagnética con la materia y la involucración de glicosilación de proteínas en señalización)
Biomedicina (el diseño y descubrimiento de fármacos, la identificación y validación de dianas farmacológicsa, y la interacción proteína-ADN)
Biología (epidemiología, evolución y las redes complejas)
El área de Biofísica en BIFI abarca 9 líneas de investigación:
- Plegamiento de proteínas y diseño molecular
- Interacciones biomoleculares
- La glicosilación de proteínas y su papel en enfermedad
- Flavoenzimas: mecanismos de acción y biotecnología
- Malplegamiento y agregación amiloide de proteínas
- Diagnóstico clínico y drug delivery
- Transducción de señales y terapias en proteínas de membrana
- Biología estructural de receptores de membrana neuronales
- Modulación enzimática y mecanismos de reacción
- Plegamiento de proteínas y diseño molecular
- Interacciones biomoleculares
- La glicosilación de proteínas y su papel en enfermedad
- Flavoenzimas: mecanismos de acción y biotecnología
- Malplegamiento y agregación amiloide de proteínas
- Diagnóstico clínico y drug delivery
- Biología estructural de receptores de membrana neuronales
- Transducción de señales y terapias en proteínas de membrana
- Modulación enzimática y mecanismos de reacción
Plegamiento de Proteínas y Diseño Molecular
Responsable de la Línea de Investigación:
Javier Sancho
Investigadores:
Dr. Juan José Galano Frutos
Dr. Helena García Cebollada
Ritwik Maity
Patricia Bruñen Fau
Dr. Verónica Iguarbe Montalbán
Darío Bazco Marco
Antonio Hidalgo Toledo
Alfonso López
RESUMEN
ESTABILIZACIÓN, INGENIERÍA Y SIMULACIÓN DE PROTEÍNAS
 Buscamos una comprensión cuantitativa de la termodinámica de las proteínas que nos permita gobernar la composición de los conjuntos proteicos. Mediante diversas técnicas biofísicas, determinamos las magnitudes termodinámicas esenciales del equilibrio de plegamiento de proteínas (ΔG, ΔH, ΔS y ΔCp) en proteínas silvestres y en variantes obtenidas mediante técnicas de ingeniería de proteínas.
Buscamos una comprensión cuantitativa de la termodinámica de las proteínas que nos permita gobernar la composición de los conjuntos proteicos. Mediante diversas técnicas biofísicas, determinamos las magnitudes termodinámicas esenciales del equilibrio de plegamiento de proteínas (ΔG, ΔH, ΔS y ΔCp) en proteínas silvestres y en variantes obtenidas mediante técnicas de ingeniería de proteínas.
Analizamos simulaciones de Dinámica Molecular para aumentar nuestra comprensión de los principios básicos. Intentamos comprender la contribución de las interacciones fundamentales (por ejemplo, diferentes tipos de interacciones electrostáticas), pero también de las entidades químicas (cadenas laterales de residuos de aminoácidos) a la estabilidad de las proteínas. Aspiramos a proporcionar herramientas conceptuales y computacionales que hagan posible un diseño verdaderamente cuantitativo de la estabilidad de las proteínas.
HTS, ANTIMICROBIANOS, CHAPERONAS FARMACOLÓGICAS, QUÍMICA MÉDICA
 Utilizamos nuestros conocimientos para desarrollar fármacos dirigidos a proteínas, ya sean humanas o de otro tipo. Podemos partir de una diana proteínica definida, identificar los «hits» y convertirlos en «leads» utilizando un enfoque completo de química médica.
Utilizamos nuestros conocimientos para desarrollar fármacos dirigidos a proteínas, ya sean humanas o de otro tipo. Podemos partir de una diana proteínica definida, identificar los «hits» y convertirlos en «leads» utilizando un enfoque completo de química médica.
Nuestros proyectos más avanzados están relacionados con nuevos antimicrobianos específicos contra Helicobacter pylori, chaperonas farmacológicas personalizadas para la fenilcetonuria y nuevos inhibidores de la agregación para la enfermedad de Parkinson.
BIOINFORMÁTICA, PREDICTORES, SIMULACIONES MD, VARIOMA HUMANO
Estamos desarrollando técnicas para aumentar la precisión de la interpretación fenotípica de las variantes genéticas de un solo nucleótido. Intentamos potenciar el uso de técnicas de MD para clasificar con precisión las variantes genéticas en benignas y deletéreas. Nos centramos en las variantes sin sentido que afectan a regiones que codifican proteínas con una función bien definida.
Entre otros, trabajamos en la interpretación de variantes en genes relacionados con enfermedades detectadas en el recién nacido.
PUBLICACIONES RELEVANTES
1.-Galano-Frutos, Juan José; Nerín-Fonz, Francho; Sancho, Javier. 2023. Calculation of Protein Folding Thermodynamics using Molecular Dynamics Simulations. J. Chem. Inf. Model. In Press
2. Galano-Frutos, Juan José, Torreblanca Renzo, García-Cebollada Helena, Sancho Javier. 2022. A look at the face of the molten globule: structural model of the Helicobacter pylori apoflavodoxin ensemble at acidic pH. Protein Science 2022,31:e4445
3. Galano-Frutos, Juan José; García-Cebollada, Helena; Sancho, Javier. 2021. Molecular Dynamics Simulations for Genetic Interpretation in Protein Coding Regions: Where we Are, Where to Go and When. Briefings in bioinformatics. 22:3–192.
4. Pujols, J.; et al. (18/14) 2018. Small molecule inhibits alpha-synuclein aggregation, disrupts amyloid fibrils, and prevents degeneration of dopaminergic neurons. Proceedings of the national academy of sciences. 115-41, pp.10481-10486. ISSN 0027-8424.
5. Espinosa Angarica, Vladimir; Orozco, Modesto; Sancho, Javier. 2016. Exploring the complete mutational space of the LDL receptor LA5 domain using molecular dynamics: Linking snps with disease phenotypes in familial hypercholesterolemia. Human molecular genetics. 25-6, pp.1233-1246. ISSN 0964-6906.
PRINCIPALES PROYECTOS DE INVESTIGACIÓN
1.MOlecular-Scale Biophysics Research Infrastructure MOSBRI. UE (H2020-INFRAIA-2020-1). 2021-2025. IP: Javier Sancho (Coordinator: Patrick England, Institute Pasteur, France)
2. Estabilizador automático de proteínas para biotecnología y biomedicina. Ministerio de Ciencia e Innovación. 2021-2024. IP: Javier Sancho
3. La estabilidad de proteínas: análisis en profundidad, nuevas herramientas de predicción y aplicaciones en biomedicina y biotecnología. PID2022-141068NB-I00. Ministerio de Ciencia e Innovación. 2023-2026. IP: Javier Sancho
Colaboradores
Mantenemos colaboraciones activas con muchos grupos nacionales e internacionales y redes: INPEC. Ver publicaciones. Ver, así mismo, las webs de proyectos: RedMut, Pirepred
CONTACTO
Interacciones Biomoleculares
Responsable de la Línea de Investigación:
Adrian Velazquez-Campoy
Investigadores:
José Luis Neira (Universidad Miguel Hernández, Elche)
Bruno Rizzuti (Institute of Nanotechnology, CNR, Italia)
David Ortega Alarcón
Ana Jiménez Alesanco
Paula García Franco
Ana Jiménez Alesanco
RESUMEN
Todos los procesos biológicos se pueden describir como una secuencia de procesos de interacción entre biomoléculas y cambios conformacionales acoplados. Nosotros trabajamos en tres cuestiones fundamentales, desde una perspectiva tanto básica como aplicada, relacionadas con interacciones en moléculas biológicas:
- Caracterización biofísica de proteínas de relevancia biotecnológica y/o biomédica: paisaje conformacional y funcional, interacciones con otras biomoléculas, cambios conformacionales, diagramas de fase, fenómenos cooperativos…
- Desarrollo de metodologías experimentales para el estudio de interacciones biomoleculares, fenómenos cooperativos y regulación funcional en sistemas biológicos.
- Desarrollo e implementación de procedimientos de cribado de alto rendimiento para la identificación de compuestos bioactivos capaces de modular la función de dianas proteicas farmacológicas.
PUBLICACIONES RELEVANTES
1. N. Losada-Garcia, A. Vazquez-Calvo, D. Ortega-Alarcon, O. Abian, A. Velazquez-Campoy, P. Domingo-Calap, A. Alcami, J.M. Palomo. Nanostructured biohybrid material with wide-ranging antiviral action. Nano Research 2023, 16:11455-11463
2.- S. Araujo-Abad, B. Rizzuti, A. Villamarin-Ortiz, D. Pantoja-Uceda, C.M. Moreno-Gonzalez, O. Abian, A. Velazquez-Campoy, J.L. Neira, C. de Juan Romero. New insights into cancer: MDM2 binds to the citrullinating enzyme PADI4. Protein Science 2023, 32:e4723
3.– S. Araujo-Abad, M. Fuentes-Baile, B. Rizzuti, J.F. Bazan, A. Villamarin-Ortiz, M. Saceda, E. Fernandez, M. Vidal, O. Abian, A. Velazquez-Campoy, C. de Juan Romero, J.L. Neira. The intrinsically disordered, epigenetic factor RYBP binds to the citrullinating enzyme PADI4 in cancer cells. International Journal of Biological Macromolecules 2023, 246:125632
4.– M. Bastos, O. Abian, C.M. Johnson, F. Ferreira-da-Silva, S. Vega, A. Jimenez-Alesanco, D. Ortega-Alarcon, A. Velazquez-Campoy. Isothermal titration calorimetry. Nature Reviews Methods Primers 2023, 3:17
5.– D. Ortega-Alarcon, R. Claveria-Gimeno, S. Vega, O.C. Jorge-Torres, M. Esteller, O. Abian, A. Velazquez-Campoy. Unexpected thermodynamic signature for the interaction of hydroxymethylated DNA with MeCP2. International Journal of Biological Macromolecules 2023, 232:123373
6.– K. Gonzalez-Arzola, A. Diaz-Quintana, N. Bernardo-Garcia, J. Martinez-Fabregas, F. Rivero-Rodriguez, M.A. Casado-Combreras, C. Elena-Real, A. Velazquez-Cruz, S. Gil-Caballero, A. Velazquez-Campoy, E. Szulc, M.P. Gavilan, I. Ayala, R. Arranz, R.M. Rios, X. Salvatella, J.M. Valpuesta, J. Hermoso, M.A. De la Rosa, I. Diaz-Moreno. Nucleus-translocated mitochondrial cytochrome c liberates nucleophosmin-sequestered ARF tumor suppressor by changing nucleolar liquid-liquid phase separation. Nature Structural & Molecular Biology 2022, 29:1024-1036
7.– A. Jimenez-Alesanco, U. Eckhard, M. Asencio del Rio, S. Vega, T. Guevara, A. Velazquez-Campoy, F.X. Gomis-Rüth, O. Abian. Repositioning small molecule drugs as allosteric inhibitors of the BFT-3 toxin from enterotoxigenic Bacteroides fragilis. Protein Science 2022, 31:e4427
8.– D. Ortega-Alarcon, R. Claveria-Gimeno, S. Vega, O.C. Jorge-Torres, M. Esteller, O. Abian, A. Velazquez-Campoy. Stabilization effect of intrinsically disordered regions on multidomain proteins: The case of the methyl-CpG protein 2, MeCP2. Biomolecules 2021, 11:1216
9.– D. Ortega-Alarcon, R. Claveria-Gimeno, S. Vega, O.C. Jorge-Torres, M. Esteller, O. Abian, A. Velazquez-Campoy. Influence of the disordered domain structure of MeCP2 on its structural stability and dsDNA interaction. International Journal of Biological Macromolecules 2021, 175:58-66
10.– B. Rizzuti, L. Ceballos-Laita, D. Ortega-Alarcon, A. Jimenez-Alesanco, S. Vega, F. Grande, F. Conforti, O. Abian, A. Velazquez-Campoy. Sub-micromolar inhibition of SARS-CoV-2 3CLpro by natural compounds. Pharmaceuticals 2021, 14:892
11. O. Abian, D. Ortega-Alarcon, A. Jimenez-Alesanco, L. Ceballos-Laita, S. Vega, H.T. Reyburn, B. Rizzuti, A. Velazquez-Campoy. Structural stability of SARS-CoV-2 3CLpro and identification of quercetin as an inhibitor by experimental screening. International Journal of Biological Macromolecules 2020, 164:1693-1703
12.– J. Felix, K. Weinhäupl, C. Chipot, F. Dehez, A. Hessel, D.F. Gauto, C. Morlot, O. Abian, I. Gutsche, A. Velazquez-Campoy, P. Schanda, H. Fraga. Mechanism of the allosteric activation of the ClpP protease machinery by substrates and active-site inhibitors. Science Advances 2019, 5:eaaw3818
13.– P. SaNtofimia‐Castaño, Y. Xia, W. Lan, Z. Zhou, C. Huang, L. Peng, P. Soubeyran, A. Velazquez‐Campoy, O. Abian, B. Rizzuti, J.L. Neira, J. Iovanna. Ligand‐based design of a potent inhibitor of NUPR1 exerting anticancer activity via necroptosis. Journal of Clinical Investigation 2019, 129:2500-2513
14.– J.L. Neira, J. Bintz, M. Arruebo, B. Rizzuti, T. Bonacci, S. Vega, A. Lanas, A. Velazquez-Campoy, J.L. Iovanna, O. Abian. Identification of a drug targeting an intrinsically disordered protein involved in pancreatic adenocarcinoma. Scientific Reports 2017 7:39732
15.– R. Claveria-Gimeno, P.M. Lanuza, I. Morales-Chueca, O.C. Jorge, S. Vega, O. Abian, M. Esteller, A. Velazquez-Campoy. The intervening domain from MeCP2 enhances the DNA affinity of the methyl binding domain and provides an independent DNA interaction site. Scientific Reports 2017 7:41635
PRINCIPALES PROYECTOS DE INVESTIGACIÓN
1. New pharmacological strategy against zinc-dependent target proteins: Allosteric inhibitors of deacetylases HDAC8 and LpxC. Ministerio de Ciencia e Innovación (PID2021-127296OB-I00). Entidades participantes: Universidad de Zaragoza. 2022-2025. IP: Adrián Velázquez Campoy (Universidad de Zaragoza – BIFI)
2.Nuevas estrategias contra el cáncer: Inhibición de las interacciones moleculares de las proteínas deiminasas de arginina (InterPATh). Generalitat Valenciana (CIAICO/2021/135). 2022-2024. Entidades participantes: Universidad Miguel Hernández. IP: Meuri del Camino de Juan Romero – José L. Neira (Universidad Miguel Hernández
3.MOlecular-Scale Biophysics Research Infrastructure (MOSBRI). Horizon 2020 – Research and Innovation Framework Programme (H2020-INFRAIA-2020-1, 101004806, RIA). Entidades participantes: Institut Pasteur, Aarhus Universitet, Universite Libre de Bruxelles, Universita Degli Studi di Genova, Rijksuniversiteit Groningen, European Molecular Biology Laboratory, Linkopings Universitet, Kemijski Institut, Birkbeck College – University of London, Centre National de la Recherche Scientifique, Biotechnologicky Ustav AV CR, Universita degli Studi di Roma La Sapienza, Universidad de Zaragoza, Malvern Panalytical, Software 4 Science Developments. 2021-2025. IP: Patrick England (Main Proposer, Institut Pasteur), Adrian Velazquez Campoy (Proposer, Universidad de Zaragoza – BIFI)
Colaboradores
- Olga Abian (Instituto Aragones de Ciencias de la Salud and BIFI, Spain)
- Juan Ausio (University of Victoria, Canada)
- Rui Brito (Universidade de Coimbra, Portugal)
- Pierpaolo Bruscolini (Universidad de Zaragoza and BIFI, Spain)
- Jose A. Carrodeguas (Universidad de Zaragoza and BIFI, Spain)
- Irene Diaz-Moreno (Instituto de Bioquimica Vegetal y Fotosintesis – CSIC, Spain)
- Maria Fillat (Universidad de Zaragoza and BIFI, Spain)
- Marcos R. Fontes (São Paulo State University, Brazil)
- Ernesto Freire (The Johns Hopkins University, USA)
- Enrique Garcia-Hernandez (Universidad Nacional Autonoma de Mexico, Mexico)
- Ruben Martinez-Buey (Universidad de Salamanca, Spain)
- Milagros Medina (Universidad de Zaragoza and BIFI, Spain)
- Maria João Moreno (Universidade de Coimbra, Portugal)
- Arturo Muga (Universidad del Pais Vasco, Spain)
- Jose A. Navarro (Instituto de Bioquimica Vegetal y Fotosintesis – CSIC, Spain)
- Julian Pardo (Universidad de Zaragoza and IIS-Aragon, Spain)
- Santiago Ramon-Maiques (CNIO, Spain)
- David Reverter (Universidad Autonoma de Barcelona, Spain)
- Javier Sancho (Universidad de Zaragoza and BIFI, Spain)
- Jayaraman Sivaraman (National University of Singapore, Singapore)
- Maria A. Urbaneja (Universidad del Pais Vasco, Spain)
- Salvador Ventura (Universidad Autonoma de Barcelona, Spain)
CONTACTO
Adrian Velazquez-Campoy, adrianvc@unizar.es
La Glicosilación de Proteínas y su Papel en Enfermedad
Responsable de la Línea de Investigación:
Ramón Hurtado Guerrero
Investigadores:
Víctor Taleb
Billy Veloz
Irene Ginés
María Bort
Alejandro Pérez
Mattia Ghirardello
Khanh Nguyễn
RESUMEN
Nuestro grupo está interesado en el estudio de las glicosiltransferasas, glicosilhidrolasas y proteínas/módulos de unión a carbohidratos implicados en enfermedades humanas. Para ello, utilizamos la cristalografía de proteínas como la principal herramienta complementada con enzimología, estudios de inhibición, etc, con el fin de estudiar los mecanismos moleculares de las enzimas que están implicadas en la síntesis, modificación y degradación de los glicoconjugados, oligo y polisacáridos (ver las publicaciones más relevantes del grupo a continuación).
Principalmente trabajamos actualmente en la O-glicosilación de proteínas y en las glicosiltransferasas responsables de esta modificación postraduccional.
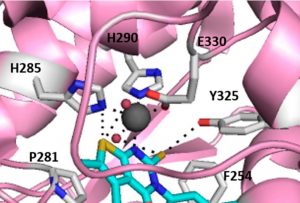
En particular, actualmente estamos trabajando en varias glicosiltransferasas como las Proteínas O-fucosiltransferasas 1 y 2 (POFUT1 y 2), FUT8 y la gran familia de GalNAc-Ts. Mientras que POFUT1 y 2 fucosilatan dominios plegados como los dominios EFG y TSR, respectivamente, GalNAc-Ts glicosilan principalmente regiones no estructuradas presentes en un gran número de proteínas como las mucinas. FUT8 es responsable de la llamada fucosilación del núcleo (Figura 1).
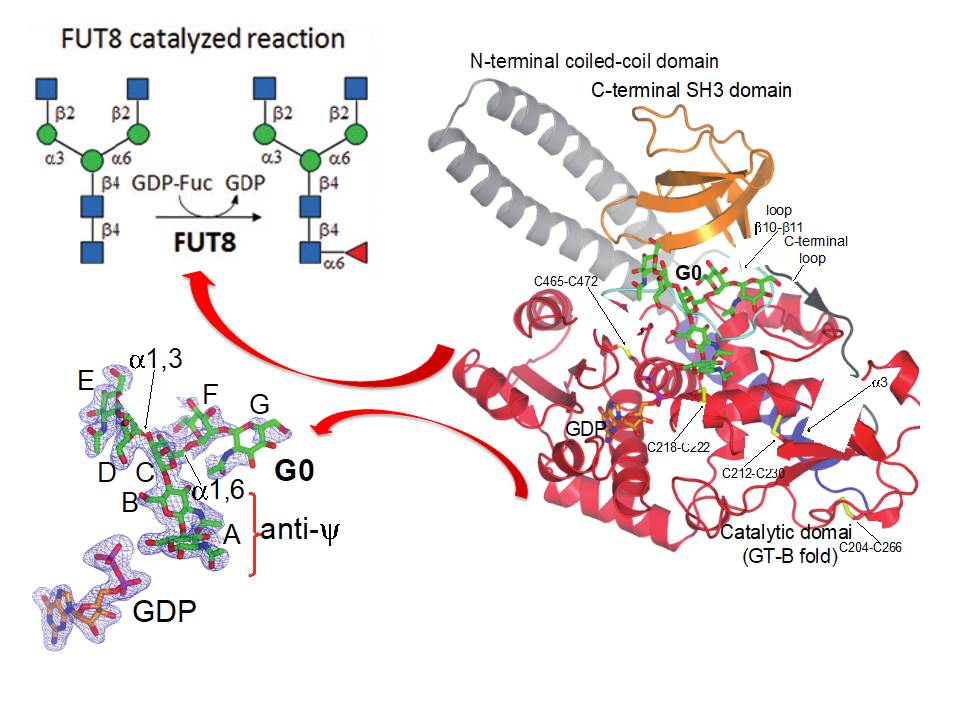
Figure 1. Scheme summarizing the structure of FUT8 complexed to GDP and a biantennary N-glycan..
Además estamos interesados en la elucidación de las coordenadas de reacción y el mecanismo catalítico mediante el uso de análogos de estado de transición o complejos de Michaelis. Finalmente estos estudios serán importantes para el diseño de nuevos fármacos con futuras aplicaciones terapéuticas frente a diversas enfermedades.
Por ultimo y aunque no relacionado con el campo de la glicobiología, tenemos una estrecha colaboración con el Prof. Dr. Guadix y Conejo-García, de la Universidad de Granada, en el desarrollo de nuevos inhibidores contra la colina quinasa a1 humana para el tratamiento del cáncer.
PUBLICACIONES RELEVANTES
1. Substrate-guided front-face reaction revealed by combined structural snapshots and metadynamics for the polypeptide GalNAc-T2. Lira-Navarrete E, Iglesias-Fernández J, Zandberg WF, Compañón I, Kong Y, Corzana F, Pinto BM, Clausen H, Peregrina JM, Vocadlo DJ, Rovira C*, Hurtado-Guerrero R*. (2014) Angew Chem Int Ed Engl 53(31):8206-10. (89 citations/9 yrs)
First trapped experimental Michaelis complex for a retaining glycosyltransferase probing the SNi mechanism
2. Dynamic interplay between catalytic and lectin domains of GalNAc-transferases modulates protein O-glycosylation. Lira-Navarrete E, de Las Rivas M, Compañón I, Pallarés MC, Kong Y, Iglesias-Fernández J, Bernardes GJ, Peregrina JM, Rovira C, Bernadó P, Bruscolini P, Clausen H, Lostao A, Corzana F, Hurtado-Guerrero R*. (2015). Nat Commun 6:6937. (92 citations/8 yrs)
First work describing how the lectin domain of GalNAc-Ts modulates O-glycosylation
3. A proactive role of water molecules in acceptor recognition by protein O-fucosyltransferase 2. Valero-González J, Leonhard-Melief C, Lira-Navarrete E, Jiménez-Osés G, Hernández-Ruiz C, Pallarés MC, Yruela I, Vasudevan D, Lostao A, Corzana F, Takeuchi H, Haltiwanger RS, Hurtado-Guerrero R*. (2016) Nat Chem Biol 12(4):240-6. (65 citations/7 yrs)
Describes the key role of water molecules in the binding of PoFUT2 to its various protein substrates
4. The interdomain flexible linker of the polypeptide GalNAc transferases dictates their long-range glycosylation preferences. de Las Rivas M, Lira-Navarrete E, Daniel EJP, Compañón I, Coelho H, Diniz A, Jiménez-Barbero J, Peregrina JM, Clausen H, Corzana F, Marcelo F, Jiménez-Osés G, Gerken TA, Hurtado-Guerrero R*. (2017) Nat Commun 8(1):1959. (35 citations/6 yrs)
First elucidation of the role of a small flexible linker dictating the long-glycosylation preference
5. Structural basis for arginine glycosylation of host substrates by bacterial effector proteins. Park JB, Kim YH, Yoo Y, Kim J, Jun SH, Cho JW, El Qaidi S, Walpole S, Monaco S, García-García AA, Wu M, Hays MP, Hurtado-Guerrero R*, Angulo J*, Hardwidge PR, Shin JS*, Cho HS*. (2018) Nat Commun 9(1):4283. (52 citations/5 yrs)
Elucidation of how arginine-glycosyltransferases from entheropathogens glycosylate human proteins to regulate immunological activity
6. Mechanisms of redundancy and specificity of the Aspergillus fumigatus Crh transglycosylases. Fang W, Sanz AB, Bartual SG, Wang B, Ferenbach AT, Farkaš V, Hurtado-Guerrero R, Arroyo J*, van Aalten DMF*. (2019). Nat Commun 10(1):1669. (21 citations/4 yrs)
Detailed study of the importance of the Crh transglycosylases in the biology of A. fumigatus
7. Structural basis for substrate specificity and catalysis of α1,6-fucosyltransferase. García-García A, Ceballos-Laita L, Serna S, Artschwager R, Reichardt NC, Corzana F, Hurtado-Guerrero R*. (2020). Nat Commun 2020;11(1):973. (37 citations/3 yrs)
Structural basis of how the fucosyltransferase FUT8 achieves core-fucosylation on N-glycans
8. Molecular basis for fibroblast growth factor 23 O-glycosylation by GalNAc-T3. de Las Rivas M, Paul Daniel EJ, Narimatsu Y, Compañón I, Kato K, Hermosilla P, Thureau A, Ceballos-Laita L, Coelho H, Bernadó P, Marcelo F, Hansen L, Maeda R, Lostao A, Corzana F, Clausen H, Gerken TA, Hurtado-Guerrero R*. (2020). Nat Chem Biol 16(3):351-360. (42 citations/3 yrs)
Describes the molecular and cellular mechanisms underlying the regulation of FGF23 by GalNAc-T3
9. Structural basis for the synthesis of the core 1 structure by C1GalT1. González-Ramírez AM, Grosso AS, Yang Z, Compañón I, Coelho H, Narimatsu Y, Clausen H, Marcelo F, Corzana F*, Hurtado-Guerrero R*. (2022) Nat Commun 13(1):2398. (6 citations/1 yr)
Molecular insights into the synthesis of core1 by C1GalT1
10. Structural and mechanistic insights into the cleavage of clustered O-glycan patches-containing glycoproteins by mucinases of the human gut. Taleb V, Liao Q, Narimatsu Y, García-García A, Compañón I, Borges RJ, González-Ramírez AM, Corzana F, Clausen H, Rovira C*, Hurtado-Guerrero R*. (2022) Nat Commun 13(1):4324. (7 citations/1 yr)
Molecular insights into AM0627 function from X-ray crystallography and computer simulations
11. Molecular basis for bacterial N-glycosylation by a soluble HMW1C-like N-glycosyltransferase. Piniello B, Macías-León J, Miyazaki S, García-García A, Compañón I, Ghirardello M, Taleb V, Veloz B, Corzana F, Miyagawa A, Rovira C*, Hurtado-Guerrero R*. (2023) Nat Commun 14(1):5785.
Structural and Molecular insights into the N-glycosylation by a soluble N-glycosyltransferase
12. Identification of global inhibitors of cellular glycosylation. Sørensen DM, Büll C, Madsen TD, Lira-Navarrete E, Clausen TM, Clark AE, Garretson AF, Karlsson R, Pijnenborg JFA, Yin X, Miller RL, Chanda SK, Boltje TJ, Schjoldager KT, Vakhrushev SY, Halim A, Esko JD, Carlin AF, Hurtado-Guerrero R, Weigert R, Clausen H, Narimatsu Y. (2023) . Nat Commun. 14(1):948.
Discovery of inhibitors affecting several pathways of glycosylation.
ACTUALES PROYECTOS DE INVESTIGACIÓN
1. PID2019-105451GB-I00.
-
- (AEI, Spain), 2020-2023. 266,200 euros plus extra funding for one PhD student. PI: Ramón Hurtado-Guerrero
2. Desarrollo de nuevas inmunoterapias para el tratamiento del cáncer y candidiasis. LMP10_21 (2021-2023). DGA. PI: Ramón Hurtado-Guerrero. 54,500 euros.
3. Nuevos tratamientos de inmunoterapia (Nanobodies y células CAR) frente a infecciones fúngicas invasivas en pacientes oncopediátricos. Convocatoria de Ayudas a la investigación del cáncer infantil. Aspanoa. 2022-2023. IP: Eva Gálvez and Maykel Arias. coIPs: Ramón Hurtado-Guerrero and Julián Pardo. 60.000 euros
4. Targeting GALNT7 to develop new drugs for the personalised treatment of prostate cancer. Prostate Cancer UK. PIs: Jennifer Munkley and Ben Schumann. Collaborators: Ramón Hurtado-Guerrero and others. 2022-2025.
5. GlyCanDrug. European Commission (ITN), HORIZON-MSCA-2022-DN-01. 251,971.20 to my group, 2024-2027. Total to the ITN = 2,696,457.60
6. PID2022-136362NB-I00 (AEI, Spain), 2023-2026. 375,000 euros plus extra funding for one PhD student. PI: Ramón Hurtado-Guerrero
Colaboradores
- Robert Haltiwanger, The University of Georgia
- Henrik Clausen, University of Copenhagen
- Daan van Aalten, University of Dundee
- Philip Hardwidge, Kansas State University
- Tom Gerken, Case Western Reserver University
- Pedro Merino, Universidad de Zaragoza
- Francisco Corzana, Universidad de La Rioja
- Carme Rovira, Universidad de Barcelona
- Filipa Marcelo, New University of Lisbon
- Julián Pardo, Universidad de Zaragoza
- Anabel Lostao, Universidad de Zaragoza
Flavoenzimas: Mecanismos de Acción y Biotecnología
Responsable de la Línea de Investigación:
Dr. Patricia Ferreira Neila
Dr. Marta Martínez Júlvez
Investigadores:
Dr. Milagros Medina Trullenque
Dr. Patricia Ferreira Neila
Dr. Marta Martínez Júlvez
Sergio Boneta Martinez
Andrea Moreno Maldonado
Olga Arjona Soriano
Maribel Bernabé Rivero
Diego Boj Carballo
Paula Cinca Fernando
Víctor Correa Pérez
Miguel Ferrer Navarro
Aurora Victoria Vázquez Rodríguez
RESUMEN
Los cofactores mononucleótido de flavina (FMN) y dinucleótido de flavina y adenina (FAD) permiten que flavoproteínas y flavoenzimas exhiban una versatilidad redox significativa en una amplia gama de procesos fisiológicos catalíticos, metabolismos primarios y secundarios, así como interacción con otras biomoléculas. Además, entre las aproximadamente 400 diferentes flavoproteínas identificadas, casi el 10% actúan en reacciones no relacionadas con procesos redox, o actúan como moléculas de señalización y detección. Algunas flavoenzimas, pueden funcionar también como potentes biocatalizadores debido a la selectividad, el control y la eficiencia en las reacciones en las que participan, mientras que otras pueden servir como dianas terapéuticas en el tratamiento de enfermedades infecciosas y trastornos patológicos en mamíferos. Sin embargo, la gran diversidad de estas proteínas y enzimas, sus características específicas de especies, así como la interacción entre ellas, con otras proteínas y diversos metabolitos, muestran que todavía estamos lejos de comprender cómo explotar todo su potencial.
En este contexto, el objetivo principal de nuestro equipo es proporcionar una mejor comprensión de los mecanismos moleculares de enzimas metabólicas clave, con especial énfasis en sistemas dependientes de flavoenzimas. Esto ayudará a clarificar sus funciones en los procesos metabólicos y redes de interacción en los que participan, así como a establecer las bases moleculares para facilitar su aplicabilidad como dianas terapéuticas o biocatalizadores.
Nuestros principales temas de investigación incluyen:
- Desentrañar las bases moleculares que determinan las funciones celulares de flavoenzimas humanas implicadas en la bioenergética celular, la señalización o la apoptosis tanto en escenarios que implican a células sanas como en situaciones que causan patologías. Este conocimiento, permitirá el desarrollo de nuevas estrategias terapéuticas para paliar el efecto de las mutaciones patógenas.
- Identificar el flavoproteoma en especies relevantes e investigar el mecanismo de acción de sus sistemas dependientes de flavoproteínas y flavoenzimas, para permitir su uso como dianas en la búsqueda de antimicrobianos y/o nuevas herramientas biotecnológicas.
- Comprender los mecanismos de incorporación del cofactor flavínico en las apoproteínas-cliente y el impacto de las modificaciones endógenas de la flavina en la funcionalidad de las flavoproteínas.
- Desarrollo de un novedoso catálogo de biocatalizadores para ser aplicados en química sintética sostenible a la producción de compuestos de base biológica de interés.
- Desentrañar la contribución de los procesos de unión y dinámica durante los mecanismos de acción de los sistemas estudiados mediante la combinación de enfoques estructurales, computacionales y experimentales.
En nuestra investigación, combinamos un gran número de herramientas bioquímicas, biofísicas, de biología molecular y biología estructural junto con enfoques computacionales para la expresión, purificación, diseño racional y caracterización estructural-funcional de proteínas y sus redes de interacción.
Las principales metodologías utilizadas en nuestra investigación incluyen:
- Producción de proteínas nativas y mutantes mediante técnicas de ingeniería de proteínas.
- Expresión de proteínas homólogas y heterólogas en diferentes microorganismos.
- Purificación de proteínas y separación de metabolitos (electroforesis, métodos cromatográficos por FPLC y HPLC, …).
- Espectrometría de absorción: estudios cinéticos en estado estacionario, espectroscopia diferencial, afinidad de unión, determinación de potenciales de reducción de punto medio.
- Cinética transitoria mediante espectroscopia de flujo detenido multimezcla.
- Ensayos de interacción y cinética en condiciones anaeróbicas.
- Espectroscopia de fluorescencia y dicroísmo circular.
- Cristalización de proteínas, difracción de rayos X y cristalografía de femtosegundos en serie (SFX) con resolución temporal de inicio y láseres de electrones libres de rayos X (XFEL).
- Técnicas avanzadas de Resonancia Paramagnética Electrónica (ESEEM, HYSCORE, ENDOR).
- Calorimetría de titulación isotérmica y calorimetría diferencial de barrido.
- Cribado de alto rendimiento de bibliotecas de compuestos químicos.
- Métodos de Biología Computacional: Acoplamiento molecular, Dinámica Molecular y simulaciones QM/MM.
- Herramientas bioinformáticas para la identificación y selección de dianas proteicas.
PUBLICACIONES RELEVANTES
1. Increased demand for FAD synthesis in differentiated and stem pancreatic cancer cells is accomplished by modulating FLAD1 gene expression: the inhibitory effect of Chicago Sky Blue. Nisco A, Carvalho TMA, Tolomeo M, Di Molfetta D, Leone P, Galluccio M, Medina M, Indiveri C, Reshkin SJ, Cardone RA, Barile M. FEBS J. 2023 Oct;290(19):4679-4694. doi: 10.1111/febs.16881
2. Beyond a platform protein for the degradosome assembly: The Apoptosis-Inducing Factor as an efficient nuclease involved in chromatinolysis. 2023. Novo N, Romero-Tamayo S, Marcuello C, Boneta S, Blasco-Machin I, Velázquez-Campoy A, Villanueva R, Moreno-Loshuertos R, Lostao A, Medina M, Ferreira P. PNAS Nexus. 2022 26;2(2):pgac312. doi: 10.1093/pnasnexus/pgac312. eCollection 2023 Feb.
3. Riboflavin kinase and pyridoxine 5′-phosphate oxidase complex formation envisages transient interactions for FMN cofactor delivery. 2023. Rivero M, Boneta S, Novo N, Velázquez-Campoy A, Polo V, Medina M. Front Mol Biosci. 10:1167348. doi: 10.3389/fmolb.2023.1167348. eCollection 2023.
4. Expanding the Physiological Role of Aryl-Alcohol Flavooxidases as Quinone Reductases. 2023. Ferreira P, Carro J. Balcells B, Martinez AT, Serrano A. Applied and Environmental Microbiology. May 31;89(5):e0184422. doi: 10.1128/aem.01844-22.
5. Counterintuitive structural and functional effects due to naturally occurring mutations targeting the active site of the disease-associated NQO1 enzyme. 2023. Pacheco-García JL, Anoz-Carbonell E, Loginov DS, Kavan D, Salido E, Man P, Medina M, Pey AL. FEBS J. 2023 Apr;290(7):1855-1873. doi: 10.1111/febs.16677.
6. A global phylogenomic analysis of the shiitake genus Lentinula. Sierra-Patev S, Byoungnam M, Naranjo-Ortiz M, Looney B, Konkel Z, Slot JC, Sakamoto Y, Steenwyk JL, Rokas A, Carro J, Camarero S, Ferreira P, … Hibbett D. 2023. PNAS. 120(10):e2214076120. doi: 10.1073/pnas.2214076120.
7. Mining the Flavoproteome of Brucella ovis, the Brucellosis: Causing Agent in Ovis aries. 2022. Minjárez-Sáenz M, Martínez-Júlvez M, Yruela I, Medina M. Microbiol Spectr. 10(2):e0229421. doi: 10.1128/spectrum.02294-21.
8. Cofactors and pathogens: FMN and FAD biosynthesis by the FAD synthase from Brucella ovis. 2022. Moreno SA, Taleb V, Sebastián M, Anoz-Carbonell EA, Martínez-Júlvez M, Medina M. IUBMB LIFE. 74:655-671. doi: 10.1002/iub.2576.
9. Atomic Force Microscopy to Elicit Conformational Transitions of Ferredoxin-Dependent Flavin Thioredoxin Reductases. 2021. Marcuello C, Frempong GA, Balsera M, Medina M, Lostao A. Antioxidants 9;10(9):1437. doi: 10.3390/antiox10091437.
10. Structural basis of the pleiotropic and specific phenotypic consequences of missense mutations in the multifunctional NAD(P)H:quinone oxidoreductase 1 and their pharmacological rescue. 2021. Pacheco-Garcia JL, Anoz-Carbonell E, Vankova P, Kannan A, Palomino-Morales R, Mesa-Torres N, Salido E, Man P, Medina M, Naganathan AN, Pey AL. Redox Biol. 46:102112. doi: 10.1016/j.redox.2021.102112.
11. W196 and the β-Hairpin Motif Modulate the Redox Switch of conformation and the Biomolecular Interaction Network of the Apoptosis-Inducing Factor. 2021. Romero-Tamayo S, Laplaza R, Velázquez-Campoy A, Villanueva R, Medina M, P. Ferreira P. Oxid Med Cell Longev. 15;2021:6673661. doi: 10.1155/2021/6673661. eCollection 2021.
12. Unexpected diversity of ferredoxin-dependent thioredoxin reductases in cyanobacteria. Buey RM, Fernández-Justel D, González-Holgado G, Martínez-Júlvez M, González-López A, Velázquez-Campoy A, Medina M, Buchanan BB, Balsera M. Plant Physiol. 2021 May 27;186(1):285-296. doi: 10.1093/plphys/kiab072.
13. Anaerobic Stopped-Flow Spectrophotometry with Photodiode Array Detection in the Presteady State: An Application to Elucidate Oxidoreduction Mechanisms in Flavoproteins Ferreira P.; Medina M. 2021. METHODS IN MOLECULAR BIOLOGY. 2280: 135-155. doi: 10.1007/978-1-0716-1286-6_9
14. The Catalytic Cycle of the Antioxidant and Cancer-Associated Human NQO1 Enzyme: Hydride Transfer, Conformational Dynamics and Functional Cooperativity. Anoz-Carbonell E, Timson DJ, Pey AL, Medina M. Antioxidants (Basel). 2020 Aug 20;9(9):772. doi: 10.3390/antiox9090772
15. Redox-and ligand binding-dependent conformational ensembles in the human apoptosis-inducing factor regulate its pro-life and cell death functions. Villanueva R.; Romero-Tamayo S.; Laplaza R.; Martinez-Olivan J.; Velazquez-Campoy A.; Sancho J.; Ferreira P*; Medina M*.. 2019. ANTIOXIDANTS & REDOX SIGNALING. 30 (18): 2013-2029. doi: 10.1089/ars.2018.7658.
PRINCIPALES PROYECTOS
1.Flavin dependent systems: multitasking roles from versatile redox catalysis to cell signaling and sensing molecules. PID2022-136369NB-I00. 2023-2026. Agencia Estatal de Investigación. IP1/2: Milagros Medina/Patricia Ferreira.
2. Grupo Referencia Biología Estructural. E35_23R. 2023-2025. Gobierno de Aragón. IP1/2: Marta Martínez Júlvez/Teresa Bes Fustero.
3. Identification and synthetic applications of novel oxidative biocatalysts in industrial biotechnology. TED2021-130803B-I00. 2022-2024. Agencia Estatal de Investigación. IP1/2: Patricia Ferreira/Juan Mangas.
4. Flavoenzymes in health, disease and drug discovery. PID2019-103901GB-I00. 2020-2023. Agencia Estatal de Investigación. IP1/2: Milagros Medina/Patricia Ferreira.
5. Sistemas Enfermedades mitocondriales asociadas al mecanismo de importación y plegamiento oxidativo de proteínas en el espacio intermembrana de la mitocondria. Mecanismos moleculares y desarrollo de nuevas estrategias terapéuticas (EMPLOXPRO). LMP27_21. Dirección General de Investigación, Gobierno de Aragón. PI: P. Ferreira. 2021-2023. IP: Ferreira P
6. El flavoproteoma de Brucella: una herramienta para dianas terapéuticas y diagnósticas. LMP13_21. Dirección General de Investigación, Gobierno de Aragón. 2021-2023. IP: Medina M.
7. Paramagnetic Species in Catalysis Research. A Unified Approach Towards Heterogeneous; Homogeneous and Enzyme Catalysis (H2020 GA Number-813209). Unión Europea. Enero 2019- Diciembre 2023. IP: Dra. García I.
8. Flavoenzimas: mecanismos y dianas moleculares; patologías y aplicaciones biotecnológicas. Ministerio de Economía y Competitividad. BIO2016-75183-P. Diciembre 2016-Diciembre 2019. IP: Dra. Medina M.
PATENTS
Enzymatic Composition and Enzymatic process for the production of 2,5-furandicarboxylic acid from 5-methoxymethylfurfural using said enzymatic composition. J. Carro, E. Fernández-Fueyo, M. Alcalde, P. Ferreira, R. Ullrich, M. Hofrichter, AT. Martinez. CSIC, University of Zaragoza and Technical University of Dresden. 2017.
COLABORADORES
Del BIFI
Dr. José Antonio Ainsa.
Dr. Patricio Fernández-Silva
Dra. Raquel Moreno Loshuertos.
Dr. Victor Polo.
Dr. Adrián Velázquez-Campoy.
Dr. Javier Sancho
De otras instituciones
Instituto de Nanociencia y Materiales de Aragón (INMA), Zaragoza
Dr. Jesús I. Martínez, Dra. Inés García-Rúbio, Dra Anabel Gracia Lostao.
Centro de Investigaciones Biológicas, CSIC, Madrid
Dr. Ángel Martínez.
Universitat degli Studi di Bari, Bari, Italia
Dra. Maria Barile.
Universidad de Granada, Granada
Dr. Ángel Pey.
Instituto de Recursos Naturales y Agrobiología de Salamanca, CSIC, Salamanca
Dra. Mónica Balsera.
Hospital Universitario Miguel Servet, Zaragoza.
Dr. M. Dolores Miramar, Dr. José Luis Caplabo.
Estación Experimental de Aula Dei
Dr. Manuel Becana, Dra. Inmaculada Yruela.
Centro de Investigación y Tecnología Agroalimentaria de Aragón
Dr. Pilar María Muñoz Álvaro.
Universitat Jaume I, Castellón
Dr. Vicent Moliner.
Universidad de Oviedo, Oviedo
Dr. Juan Mangas-Sanchez.
IIS Hospital 12 de Octubre, Madrid
Dr. María Morán
Universita degli Studi di Milano, Italia
Dr. Alessandro Aliverti.
Mitchell Cancer Institute, University of South Alabama, USA
Dra. Marie Migaud.
Instituto Química-Física Blas Cabrera, Madrid
Dr. José Manuel Martin.
Universita degli Studi di Torino, Turín, Italia
Dr. Salvatore Adinolfi, Dra. Sheila Sadeghi.
CONTACTO
Patricia Ferreira, ferreira@unizar.es, https://orcid.org/0000-0003-4076-6118
Marta Martínez Júlvez, mmartine@unizar.es, https://orcid.org/0000-0001-9047-0046
Milagros Medina, mmedina@unizar.es, https://orcid.org/0000-0001-8743-0182
Malplegamiento y Agregación Amiloide de Proteínas
Responsable de la Línea de Investigación:
Dr. Nunilo Cremades
Investigadores:
Nunilo Cremades
José Daniel Camino Camino
David Polanco Irisarri
Alejandra Carrancho Arroyo
María Martínez Monge
Blanca Viguri Lamata
RESUMEN
El fenómeno de malplegamiento y agregación amiloide de proteínas ha emergido en los últimos años como un tema de relevancia fundamental en un amplio rango de disciplinas científicas como la física, química, biología y medicina. Esta explosión de interés ha surgido tras el reconocimiento de que aproximadamente 50 enfermedades y desordenes humanos están asociados con el proceso de agregación amiloide de proteínas, algunos de los cuales se encuentran entre las condiciones médicas más comunes y debilitantes del mundo moderno, incluyendo la enfermedad de Alzheimer, de Parkinson y la diabetes tipo II.
A pesar del impacto social y económico de algunas de estas enfermedades, se desconocen los orígenes moleculares y mecanismos de la agregación amiloide de proteínas, así como de su toxicidad asociada. En los últimos años, se ha propuesto que procesos aberrantes de separación de fases líquido-líquido y líquid-sólido de proteínas puedan estar implicados en los eventos iniciales de la formación de agregados amiloides en muchas de estas enfermedades. La investigación desarrollada en el grupo liderado por la Dra. Nunilo Cremades aspira a resolver estas cuestiones fundamentales mediante la combinación de un amplio rango de técnicas biofísicas, incluyendo fluorescencia de partícula única, con experimentos de biología celular. Se pretende, además, identificar nuevas dianas proteicas para el desarrollo de herramientas de diagnóstico precoz y de tratamientos más efectivos para este tipo de enfermedades.
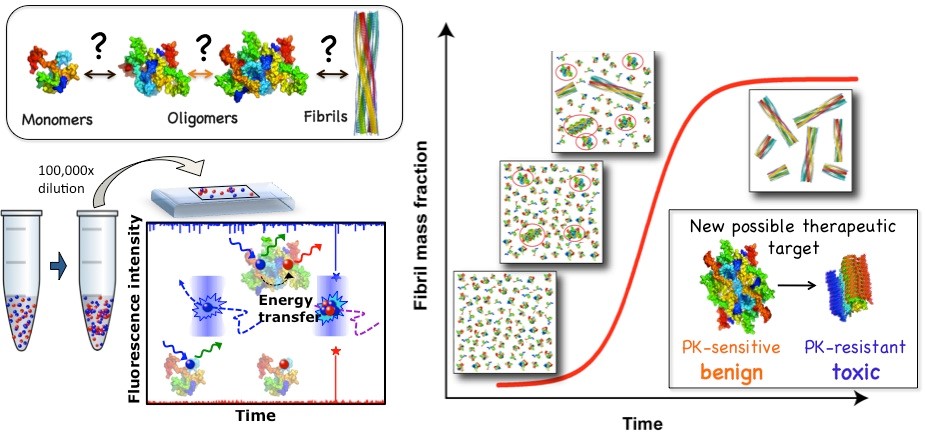
Figura 1. El desarrollo y la aplicación de técnicas de fluorescencia de partícula única nos ha permitido investigar el proceso de agregación amiloide con un nivel de detalle sin precedente e identificar nuevas posibles dianas terapéuticas. Cremades N. et al. Cell 2012.
Figura 2. Hemos sido capaces de purificar especies oligoméricas muy estables de alfa-synucleina, proteína cuya agregación y deposición está asociada al desarrollo de la enfermedad de Parkinson, y hemos mostrado que estas especies son altamente tóxicas y tienen propiedades generales comunes con otras especies oligoméricas generadas por otras proteínas amiloidogénicas. Mediante la combinación de un amplio rango de métodos biofísicos con técnicas de reconstrucción de imágenes de criomicroscopía electrónica hemos sido capaces de obtener modelos estructural tridimensionales y revelar la arquitectura estructural cuaternaria de estos oligoméros tóxicos de alfa-synucleina. Chen SW. et al. PNAS USA 2015; Fusco G et al. Science 2017; Cascella R et al. Nat. Commun. 2021
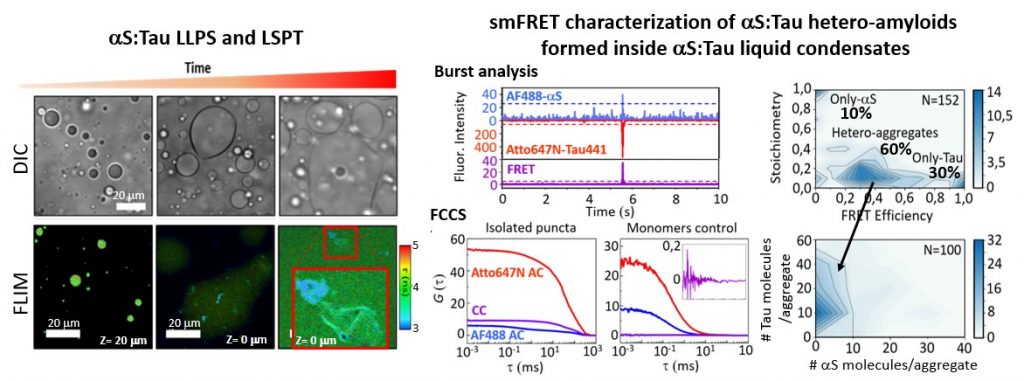
Figura 3. Hemos identificado que las proteínas alfa-sinucleina (aS) y Tau son capaces de formar condensados líquidos por un proceso de separación de fases líquido-líquido (LLPS) y que con el tiempo se forman agregados amiloides que contienen ambas proteínas en su interior. Este proceso podría estar involucrado en la formación de hetero-agregados amiloides de ambas proteínas encontrados en el cerebro de pacientes de Parkinson. Hemos puesto a punto una batería de técnicas de fluorescencia para estudiar procesos de LLPS y LSPT (liquid-to-solid phase transitions) como FLIM, FCCS, smFRET. Gracia P. and Polanco D. et al. Nat. Commun. 2022
PUBLICACIONES RELEVANTES
1. Gracia P and Polanco D, Tarancón-Díez J, Serra I, Bracci M, Oroz J, Laurents DV, Gracía I, Cremades N*. Molecular mechanism for the synchronized electrostatic coacervation and co-aggregation of alpha-synuclein and Tau. Nat. Commun. (2022). Aug 6;13(1):4586. doi: 10.1038/s41467-022-32350-9.
2. Santos J, Gracia P, Navarro S, Peña-Díaz S, Pujols J, Cremades N*, Pallarés I*, Ventura S*. Alpha-helical peptidic scaffolds to target alpha-synuclein toxic species with nanomolar affinity. Nat. Commun. (2021) Jun 18; 12(1):3752. doi: 10.1038/s41467-021-24039-2.
3. Cascella R, Chen SW, Bigi A, Camino JD, Xu CK, Dobson CM, Chiti F, Cremades N*, Cecchi C*. The release of toxic oligomers from alpha-synuclein fibrils induces dysfunction in neuronal cells. Nat. Commun. (2021) Mar 22; 12(1):1814. doi: 10.1038/s41467-021-21937-3.
4. Camino JD, Gracia P, Chen SW, Sot J, de la Arada I, Sebastián V, Arrondo JLR, Goñi FM, Dobson CM, Cremades N*. The extent of protein hydration dictates the preference for heterogeneous or homogeneous nucleation generating either parallel or antiparallel beta-sheet alpha-synuclein aggregates. Chem. Sci. (2020) Oct 15; 11(43):11902-11914. doi: 10.1039/d0sc05297c.
5. Froula JM, Castellana-Cruz M, Anabtawi NM, Camino JD, Chen SW, Thrasher DR, Freire J, Yazdi AA, Fleming S, Dobson CM, Kumita JR, Cremades N*, Volpicelli-Daley LA*. Defining alpha-synuclein species responsible for Parkinson´s disease phenotypes in mice. J. Biol. Chem. (2019) Jul 5; 294(27):10392-10406.
6. Fusco G, Chen S., Williamson PTF, Cascella R, Perni M, Jarvis JA, Cecchi C, Vendruscolo M, Chiti F, Cremades N*, Ying L, Donson CM*, De Simone A*. Structural basis of membrane disruption and cellular toxicity by alpha-synuclein oligomers. Science (2017) Dec 15;358(6369):1440-1443.
7. Chen SW, Drakulic S, Deas E, Ouberai M, Aprile FA, Arranz R, Ness S, Roodveldt C, Guilliams T, De-Genst EJ, Klenerman D, Wood NW, Knowles TPJ, Alfonso C, Rivas G, Abramov AY, Valpuesta JM, Dobson CM*, Cremades N*. Structural characterization of toxic oligomers that are kinetically trapped during α-synuclein fibril formation. Proc. Natl. Acad. Sci U.S.A. (2015) Apr 21;112(16):E1994-2003.
8. Cremades N, Cohen SI, Deas E, Abramov AY, Chen AY, Orte A, Sandal M, Clarke RW, Dunne P, Aprile FA, Bertoncini CW, Wood NW, Knowles TPJ, Dobson CM*, Klenerman D*. Direct observation of the interconversion of normal and toxic forms of alpha-synuclein. Cell (2012) 149(5): 1048-59.
PRINCIPALES PROYECTOS DE INVESTIGACIÓN
1. GAP-101098989. IVBM-4PAP: Development of an In-Vivo Brillouin Microscope (with application to Protein Aggregation-based Pathologies). Sponsor: European Union -HORIZON-EIC-2022-PATHFINDEROPEN-01. 2023-2026. Total granted: 3.333.513 €. To the Univ. Zaragoza: 530.198,75 €. PI: N. Cremades
2. PID2022-136997NB-I00: Understanding liquid-liquid phase separation and liquid-to-solid phase transitions in amyloidogenic proteins: New paradigms in Alzheimer´s and Parkinson´s disease. Sponsor: Agencia Estatal de Investigación. 2023-2026. 250.000 €. PI: N. Cremades
3. LMP17_21: Development and validation of biomarker assays for the early diagnosis of Parkinson´s disease. Sponsor: Gobierno de Aragón. 2021-2023. 99.948,8€. PI: N. Cremades
Colaboradores
Laura Volpicelli-Daley (University of Alabama, USA)
Fabrizio Chiti (University of Florence, Italy)
Janet Kumita (University of Cambridge, UK)
Douglas V. Laurents (IQFR-CSIC, Spain)
Felix Goñi (Basque Centre for Biophysics, Spain)
Arturo Muga (Basque Centre for Biophysics, Spain)
Paola Picotti (ETH Zurich, Switzerland)
Contacto
Dra. Nunilo Cremades
Instituto de Biocomputación y Física de Sistemas Complejos
Universidad de Zaragoza
Mariano Esquillor S/N. Edificio I+D+i
Zaragoza 50018, Spain
Teléfono: +34-876555417
Email: ncc@unizar.es
Web del grupo
https://sites.google.com/view/neuromol/p%C3%A1gina-principal
Diagnóstico Clínico y Drug Delivery
Responsable de la Línea de Investigación:
Olga Abian Franco
Investigadores:
Rafael Clavería, Predoctoral Student
María Arruebo, Predoctoral Student
Alberto Rodrigo, Predoctoral Student
Arturo Vinuesa, Predoctoral Student
RESUMEN
Diagnóstico clínico
La Calorimetría diferencial de barrido (DSC) ha surgido recientemente como una técnica prometedora que proporciona información útil sobre interactómica de suero / plasma (composición en proteínas y metabolitos, así como sus interacciones).
Como resultado de la enfermedad, la composición de suero / plasma se altera y es posible discriminar entre individuos sanos y los pacientes con ciertas enfermedades.
Un estudio previo realizado en nuestro grupo (25 sujetos sanos y 60 pacientes con adenocarcinoma gástrico) mostró que había diferencias significativas en las variables obtenidas a partir de los perfiles calorimétricos de pacientes con adenocarcinoma gástrico y sanos y también entre pacientes con diferentes estadios de la enfermedad.
En esta línea de investigación se propone la validación y la aplicación de una metodología (DIGCAL) como una herramienta útil en el diagnóstico y seguimiento de patologías tumorales (adenocarcinoma ductal pancreático, lesiones quísticas pancreáticas preneoplásicas y cáncer de estómago), así como en el aislamiento y la identificación de biomarcadores potenciales tumorales.
Los perfiles calorimétricos de suero de sujetos sanos y pacientes con los tres tipos de cáncer en diferentes etapas se obtendrán antes y después de 6 meses de tratamiento clínico. El análisis multiparamétrico de los perfiles térmicos nos permitirá establecer un protocolo clínico para: 1 / cribado de un determinado proceso tumoral; 2 / pacientes que clasifican de acuerdo a su estadio tumoral; 3 / control de la progresión o el control de la enfermedad; 4 / identificación de biomarcadores específicos para cada tipo de enfermedad.
DIGCAL podría llegar el nivel clínico no sólo como una herramienta de diagnóstica, sino también como un sistema de monitorización y seguimiento de los pacientes durante el tratamiento terapéutico, la adición de valor en las decisiones de pronóstico o farmacológicos. Al mismo tiempo, los biomarcadores potenciales identificados podrían ser parte de un kit de diagnóstico fácil de usar para los puntos de atención primaria.
Drug Delivery
Drug delivery es el método o proceso de administración de un compuesto farmacológico para alcanzar un efecto terapéutico en humanos o animales. Las tecnologías de drug delivery modifican el perfil de liberación del fármaco (absorción, distribución, y eliminación) en beneficio de la mejora de su eficacia y seguridad, además de evitar inconvenientes a los pacientes y hacer más cómodo su utilización. La liberación de las drogas puede basarse en difusión, degradación, y mecanismos de afinidad. Las rutas de administración más comunes incluyen preferentemente las vías no invasivas como peroral (a través de la boca), tópica (piel), transmucosal (nasal, bucal/sublingual, vaginal, ocular y rectal) y rutas de inhalación.
Muchos medicamentos como péptidos, proteínas, anticuerpos, vacunas y drogas basadas en genes, generalmente no pueden ser utilizados utilizando estas rutas ya que pueden ser susceptibles de degradación enzimática o puede que no sean incorporadas al sistema circulatorio a una concentración terapéuticamente efectiva debido a su tamaño molecular y carga. Por esta razón muchos fármacos basados en proteínas y péptidos han de ser distribuidos mediante inyección (por ejemplo, muchas inmunizaciones en las que han de liberarse proteínas).

Los esfuerzos actuales en el área de drug delivery incluyen el desarrollo de liberación dirigida en el que el fármaco sólo es activo en una zona concreta del cuerpo (por ejemplo, tejido cancerígeno) y las formulaciones son de liberación sostenida en las que el fármaco se libera durante un cierto periodo de tiempo de manera controlada. Para alcanzar una eficiente liberación dirigida, el sistema diseñado debe evitar los mecanismos de sistema de defensa del paciente y llegar a su pretendido sitio de acción. Tipos de formulaciones de liberación sostenida incluyen liposomas, microesferas cargadas biodegradables y polímeros conjugados con fármacos. En este sentido, las nanopartículas (NP) en biomedicina representan una tecnología prometedora para el transporte y liberación de fármacos. Hay muchas posibilidades de funcionalización de la superficie de las NP y gracias a esta versatilidad, se pueden desarrollar diferentes estrategias para incluir fármacos en ellas (permitiendo a las NP ir mayoritariamente al lugar de acción).
Esta línea de investigación representa una nueva estrategia para la inclusión de algunos compuestos antivirales activos contra el virus de la hepatitis C (VHC), que se desarrollaron previamente en este grupo.
Se han utilizado varios materiales:
1/ Ciclodextrinas:
La estructura química de las CD, oligosacáridos cíclicos compuestos de residuos alfa-1,4-glucosídicos, les confieren propiedades estructurales y propiedades físico-químicas óptimas que permiten su uso como portadores moleculares.
En su cavidad hidrofóbica pueden ser atrapados una amplia gama de compuestos que van desde los iones a las proteínas. Además, presentan baja citotoxicidad e inmunogenicidad.
Los complejos CD-droga se han utilizado en el campo farmacéutico con el fin de mejorar las propiedades de absorción, distribución, metabolismo, excreción y toxicidad (ADMET) relacionados con la PI de un fármaco (por ejemplo, solubilidad, la estabilidad, la entrega y la liberación, la permeabilidad de la membrana y la absorción, la toxicidad). En la actualidad, más de 30 productos que se pueden encontrar en el mercado basados en complejos de CD.
2/ Shell Cross-Linked Polymeric Micelles
Las micelas poliméricas reticuladas (CLPM), formadas por copolímeros de bloques anfifílicos, han tenido éxito para aplicaciones biomédicas. Primero, puedan cumplir con esos requisitos generales para los sistemas de administración de fármacos: solubilidad en agua, de baja toxicidad, para aumentar la estabilidad del fármaco dentro de los organismos vivos, para facilitar la captación celular en comparación con el fármaco libre, y para producir su liberación controlada en un lugar específico. En segundo lugar, la naturaleza anfifílica de los resultados de polímeros constituyentes en un núcleo hidrófobo y una cubierta hidrófila que permite la encapsulación de los dos tipos de drogas. En tercer lugar, estas nanopartículas ofrecen aún más la estabilidad en condiciones de alta dilución, debajo de la concentración micelar crítica, en comparación con otras micelas poliméricas.
En efecto, la reticulación evita su desintegración en el torrente sanguíneo y la liberación de la droga antes de llegar a la célula diana. En particular, la fijación de la estructura de micelas por reticulación covalente inducida por la luz, sobre todo el empleo de grupos reactivos de acrilato, representa un procedimiento limpio y eficaz para preparar micelas poliméricas estables que pueden contener cualquiera de las moléculas solubles en agua y no solubles en agua y el transporte a través de el torrente sanguíneo.
3/ Block Copolymers Micelles
Los vehículizadores de fármacos poliméricos son uno de los retos actuales de la nanomedicina. Desde que se introdujo el concepto de encapsulación de fármacos física dentro de los agregados poliméricos, se han identificado un número significativo de ellos. En particular, la construcción de vehículos de fármacos basados en copolímero de bloques anfifílicos es un tema de gran interés y estimulante desde el punto de vista de la investigación interdisciplinaria en química, biología y ciencia de los materiales. En medio acuoso, el autoensamblaje de copolímeros en bloque anfifílicos (BCS) sirve para minimizar las interacciones hidrófobas energéticamente desfavorables en agua y pueden conducir a una gran variedad de nanoestructuras poliméricas incluyendo especialmente micelas esféricas y vesículas atractivas.
PUBLICACIONES RELEVANTES
1.- Polymeric micelles from block copolymers containing 2,6-diacylaminopyridine units for encapsulation of hydrophobic drugs.
Concellón A, Clavería-Gimeno E, Velázquez-Campoy A, Abian O, Piñol M, Oriol L.RSC Advances, 2016, 6, 24066–24075.
2.- Biophysical Screening for Identifying Pharmacological Chaperones and Inhibitors against Conformational and Infectious Diseases. Velazquez-Campoy A, Sancho J, Abian O, Vega S. Curr Drug Targets. 2016 Jan 31. [Epub ahead of print]
3.- Cysteine Mutational Studies Provide Insight into a Thiol-Based Redox Switch Mechanism of Metal and DNA Binding in FurA from Anabaena sp. PCC 7120. Botello-Morte L, Pellicer S, Sein-Echaluce VC, Contreras LM, Neira JL, Abian O, Velázquez-Campoy A, Peleato ML, Fillat MF, Bes MT. Antioxid Redox Signal. 2016, 24, 173-185.
4.- On the link between conformational changes, ligand binding and heat capacity. Vega S, Abian O, Velazquez-Campoy A. Biochim Biophys Acta. 2015 Oct 14. pii: S0304-4165(15)00274-3.
5.- Shell Cross-Linked Polymeric Micelles as Camptothecin Nanocarriers for anti-HCV Therapy. Jiménez-Pardo I, González-Pastor R, Lancelot A, Claveria-Gimeno R, Velázquez-Campoy A, Abian O, Ros MB, and Sierra T. Macromol. Biosci. 2015, 15, 1381–1391.
6.- Su1990 A New Technology for the Classification of Patients With Gastric Adenocarcinoma Based on Differential Scanning Calorimetry Serum Thermograms. Vega S, Garcia-Gonzalez MA, Lanas A, Velazquez-Campoy A, Abian O.*. Gastroenterology 148(4): S-569 · April 2015
7.- Rescuing compound bioactivity in a secondary cell-based screening by using γ-cyclodextrin as a molecular carrier. Clavería-Gimeno R, Vega S, Grazu V, De la Fuente JM, Lanas A, Velazquez-Campoy A, Abian O.*. International Journal of Nanomedicine 2015, 10: 2249-2259.
8.- Deconvolution Analysis for Classifying Gastric Adenocarcinoma Patients Based on Differential Scanning Calorimetry Serum Thermograms. Vega S, Garcia-Gonzalez MA, Lanas A, Velazquez-Campoy A, Abian O.*. Scientific reports, 5:7988 (2015).
9.- Ionic liquids in water: a green and simple approach to improve activity and selectivity of lipases.
Filice M, Romero O, Abian O, De las Rivas B and Palomo J.M. RSC Advances 4: 49115- 49122 (2014).
10.- A unified framework based on the binding polynomial for characterizing biological systems by isothermal titration calorimetry. Vega S., Abian O.*and Velazquez-Campoy A. Methods. 2014 pp: S1046-2023(14) 00316-8.
11.- Allosteric Inhibitors of the NS3 Protease From the Hepatitis C Virus. Abian O.*, Vega S., Sancho J., Velazquez-Campoy A. PLOSone 2013, 8 (7): 69773.
12.- NS3 protease from hepatitis C virus: Biophysical studies on an intrinsically disordered protein domain. Vega S., Neira J.L., Marcuello C., Lostao A., Abian O. * and Velazquez-Campoy A. International Journal of Molecular Sciences 2013, 14: 13282-13306.
13.- Experimental Validation of In Silico Target Predictions on Synergistic Protein Targets. Cortes-Ciriano I, Koutsoukas A, Abian O, Velazquez-Campoy A and Bender A. MedChemComm 2013, 4, 278–288.
14.- Altering the interfacial activation mechanism of a lipase by solid-phase selective chemical modification. López-Gallego F, Abian O, Guisán JM. Biochemistry. 2012 Sep 4;51(35):7028-36.
15.- Semisynthetic peptide-lipase conjugates for improved biotransformations. Romero O, Filice M, de las Rivas B, Carrasco-Lopez C, Klett J, Morreale A, Hermoso JA, Guisan JM, Abian O, Palomo JM. Chem Commun (Camb). 2012 Sep 18;48(72):9053-5.
PRINCIPALES PROYECTOS DE INVESTIGACIÓN
Proyectos Actuales:
1.- Analysis of protein/metabolites interactions in plasma serum using calorimetry: application as a quick and noninvasive diagnostic method for early detection and monitoring of tumoral digestive diseases (DIGCAL). Funding Institution: Health Institute Carlos III. From: January 2016 To: December 2018. Principal Investigator (PI): Olga Abian Franco.
2.- Validación de un nuevo método diagnóstico en suero, rápido no invasivo para detección precoz de cáncer de páncreas (PANCal). Funding Institution: Asociación Española de Gastroenterología (AEG). From: 2015 To: 2016. Principal Investigator (PI): Olga Abian Franco.
Proyectos Anteriores:
3.- Adapted Nanoparticles for transport and specific release of drugs against hepatitis C virus (VHC).
Funding Institution: Health Institute Carlos III. From: 2011 To: 2013. Principal Investigator (PI): Olga Abian Franco.
4.- Implementation of in vitro e in vivo studies of anti-infectious compounds effective against Helicobacter pylori y el HCV (hepatitis C virus). Funding Institution: Health Institute Carlos III. From: 2008 To: 2011. Principal Investigator (PI): Olga Abian Franco.
Colaboradores
Colaboradores del BIFI
Dr. Adrián Velázquez-Campoy
Prof. Javier Sancho
Dr. Jose Luis Neira
Colaboradores de otras Instituciones
James Graham Brown Cancer Center, University of Louisville, Louisville, KY, EEUU
PhD. Nichola Garbett
Instituto Aragonés de Ciencias de la Salud (I+CS), Zaragoza
Prof. Angel Lanas
Dra. Trinidad Serrano
Dra. Estela Solanas
Instituto de Ciencia de Materiales de Aragón (ICMA). Química Orgánica. Facultad de Ciencias.
Dra. Teresa Sierra
Prof. Luis Oriol
Prof. Milagros Piñol
Instituto de Nanociencia (INA), Universidad de Zaragoza, Zaragoza
Dr. Jesús Martínez de Lafuente
Dra. Valeria Grazú
Dra. Berta Saez
Universidad de San Jorge (USJ)
Prof. Victor López
Prof. Elisa Langa
Instituto de Catálisis y Petroleoquímica, CSIC, Madrid.
Dr. Jose Miguel Palomo
Dr. Fernando López Gallego
Dr. Jose Manuel Guisan
Universidad de Zaragoza
Dr. Jose Antonio Ainsa
Unidad de Investigación Traslacional, Hospital Universitario Miguel Servet, Zaragoza
Dra. Pilar Alfonso
Dra. Pilar Giraldo
Dr. Miguel Pocovi
Servicio de Microbiología-INIBIC. Complejo Hospitalario Universitario A Coruña, La Coruña
Dr. Francisco José Pérez-Llarena
Biología estructural de receptores de membrana neuronales
Responsable de la Línea de Investigación:
Beatriz Herguedas Francés
Investigadores:
Carlos Vega Gutiérrez (PhD student)
Irene Sánchez Valls (PhD student)
Victoria del Pilar Ribón Fuster (TFM student)
RESUMEN
Los receptores de glutamato tipo AMPA (AMPAR) son canales iónicos regulados que median la neurotransmisión rápida excitatoria en el sistema nervioso central y están involucrados en la plasticidad neuronal. Su disfunción se asocia con varias enfermedades, como la esclerosis lateral amiotrófica, la epilepsia y el infarto cerebral. Los receptores AMPAR son un grupo diverso de complejos proteicos con diferentes propiedades cinéticas, de tráfico celular y farmacológica. Esta diversidad se alcanza a diferentes niveles. Por un lado, el receptor tetramérico contiene un núcleo compuesto por 4 subunidades, (GluA1-4) que se ensamblan con diferentes estequiometrías (Herguedas et al 2013). Esta composición varía en diferentes regiones cerebrales y etapas del desarrollo (Schwenk et al 2014). El splicing alternativo y la edición de RNA aumentan todavía más la diversidad estructural y funcional de las subunidades GluA (Traynellis et al 2010). Finalmente, esta heterogenidad estructural se expande gracias a la interacción con más de 30 proteínas que interaccionan tanto de manera estable como transitoria con el núcleo tetramérico de GluA. El objetivo de nuestra línea de investigación es indagar en la diversidad estructural de los diferentes complejos AMPARs, utilizando la crío-microscopía electrónica, con el fin de determinar los mecanismos de acción de los receptores. Además, estamos interesados en explorar el efecto de lípidos y pequeñas moléculas el mecanismo de acción del receptor así como en aplicar técnicas de molécula única para explorar su dinámica.
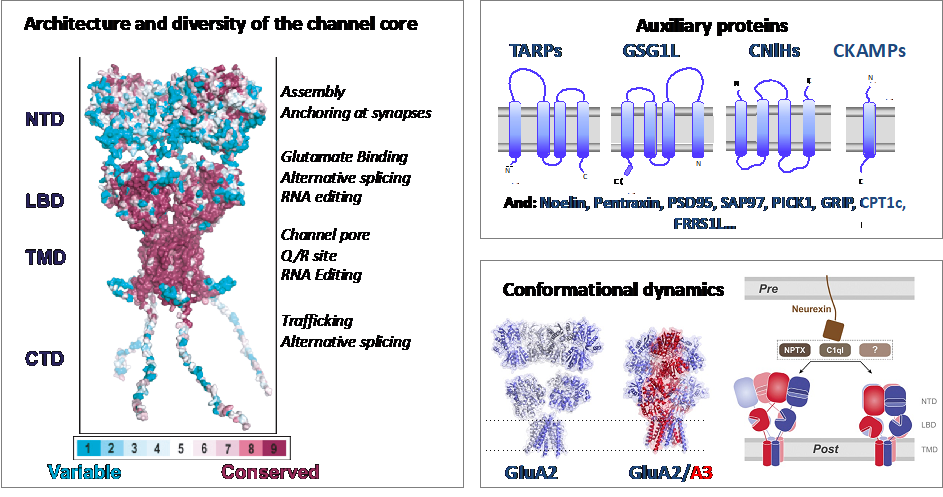
El grupo liderado por Beatriz Herguedas forma parte del grupo DGA Neuromol, junto a los IPs Jose A. Carrodeguas, Nunilo Cremades y Javier García Nafría. Este grupo tiene como objetivo caracterizar proteínas neuronales -receptores, canales iónicos y proteínas intrínsecamente desordenadas- implicadas en neuropatologías. Además, somos parte grupo de Acción BioF-DTE (Implementación y desarrollo de herramientas BIOFísicas en el estudio, Diagnóstico y Tratamiento de Enfermedades)” del campus Iberus, un grupo de acción interuniversitario y transfronterizo con participación de las Universidades de Zaragoza, Lleida, La Rioja, Navarra y Toulouse (CNRs).
PUBLICACIONES RELEVANTES
- Mechanisms underlying TARP modulation of the GluA1/2-γ8 AMPA receptor. Herguedas B, Kohegyi BK, Dohrke JN, Watson JF, Zhang D, Ho H, Shaikh SA, Lape R, Krieger JM, Greger IH. Nat Commun. 2022 Feb 8;13(1):734. doi: 10.1038/s41467-022-28404-7.
- Architecture of the heteromeric GluA1/2 AMPA receptor in complex with the auxiliary subunit TARP y8. Herguedas B*; Watson JF, Ho H; Cais O; García-Nafría J; Greger H*. Science, 2019 (*corresponding author)
- Druggability simulations of ionotropic glutamate receptors reveal a high-susceptibility binding site in the GluA3 AMPA receptor N-terminal domain. Lee* JY, Krieger J. *, Herguedas B. *, García-Nafría J. *, Dutta A., Shaikh S., Greger I.H., and Bahar I.. Structure, 2018. (*co-first author)
- Structure and organization of heteromeric AMPA-type glutamate receptors Herguedas B*, García-Nafría J*, Cais O, Fernández-Leiro R, Krieger J, Ho H, Greger IH.. Science. 2016. (*co-first author)
- The dynamic AMPA receptor extracellular region: a platform for synaptic protein interactionS. García-Nafría J, Herguedas B, Watson JF, Greger IH. s. J Physiol. Review. 2016.
- Structural insights into the synthesis of FMN in prokaryotic organisms. Herguedas B, Lans I, Sebastián M, Hermoso JA, Martínez-Júlvez M, Medina M. Acta Crystallogr D Biol Crystallogr. 2015.
- Mapping the interaction sites between AMPA receptors and TARPs reveals a role for the receptor N-terminal domain in channel gating. Cais O, Herguedas B, Krol K, Cull-Candy SG, Farrant M, Greger IH. Cell Reports, 2014.
- A hydrogen bond network in the active site of Anabaena ferredoxin-NADP(+) reductase modulates its catalytic efficiency. Sánchez-Azqueta A, Herguedas B, Hurtado-Guerrero R, Hervás M, Navarro JA, Martínez-Júlvez M, Medina M. BBA bioenergetics. 2014.
- Receptor heteromeric assembly-how it works and why it matters: the case of ionotropic glutamate receptors. Herguedas B, Krieger J, Greger IH. Prog Mol Biol Transl Sci. Review. 2013.
- Oligomeric state in the crystal structure of modular FAD synthetase provides insights into its sequential catalysis in prokaryotes Herguedas B, Martínez-Júlvez M, Frago S, Medina M and Hermoso JA.. J Mol Biol 2010.
- Crystallization and preliminary X-ray diffraction studies of FAD synthetase from Corynebacterium ammoniagenes. Herguedas B, Martínez-Júlvez M, Frago S, Medina M and Hermoso JA. Acta Crystallogr Sect F Struct Biol Cryst Commun. 2009.
- Flavodoxin: a compromise between efficiency and versatility in the electron transfer from Photosystem I to Ferredoxin-NADP(+) reductase. Goñi G, Herguedas B, Hervás M, Peregrina JR, De la Rosa MA, Gómez-Moreno C, Navarro JA, Hermoso JA, Martínez-Júlvez M, Medina M. BBA Bioenergetics. 2009.
- Protein motifs involved in coenzyme interaction and enzymatic efficiency in anabaena ferredoxin-NADP+ reductase. Peregrina JR*, Herguedas B*, Hermoso JA, Martínez-Júlvez M, Medina M Biochemistry. 2009. (*co-first author)
PRINCIPALES PROYECTOS DE INVESTIGACIÓN
- Structure and Dynamics of Calcium Permeable AMPA receptors. Agencia Estatal de Investigación. PID2019-106284GA-I00. (01/06/2020-31/05/2023). Principal Investigator.
- Dotación Adicional Programa Ramón y Cajal. Agencia Estatal de Investigación. RYC2018-025720-I. 01/05/2020-30/06/2025. Principal Investigator.
- AMPA Glutamate Receptors: the role of the extracellular domains in receptor assembly and allosteric regulation MRC Centenary Early Career Award. P.I. Beatriz Herguedas. 01/08/2012-30/09/2013. Principal Investigator.
Colaboradores
Ingo Greger (MRC Laboratory of Molecular Biology)
Javier García Nafría (Universidad de Zaragoza)
David Soto del Cerro (Universidad de Barcelona)
Raúl Estévez (Universidad de Barcelona)
CONTACTO:
bherguedas@unizar.es
https://sites.google.com/unizar.es/herguedas-lab/
Transducción de señales y terapias en proteínas de membrana
Responsable de la Línea de Investigación:
Javier García Nafría
Investigadores:
Dr. Daniel Muñoz Reyes
Dr. Josep Argerich
Sandra Arroyo Urea
Ángela Carrión Antolí
Iris del Val García
Laia Mata Rios
Adriana Tejero Dia
RESUMEN
Complejos proteicos de la membrana cerebral
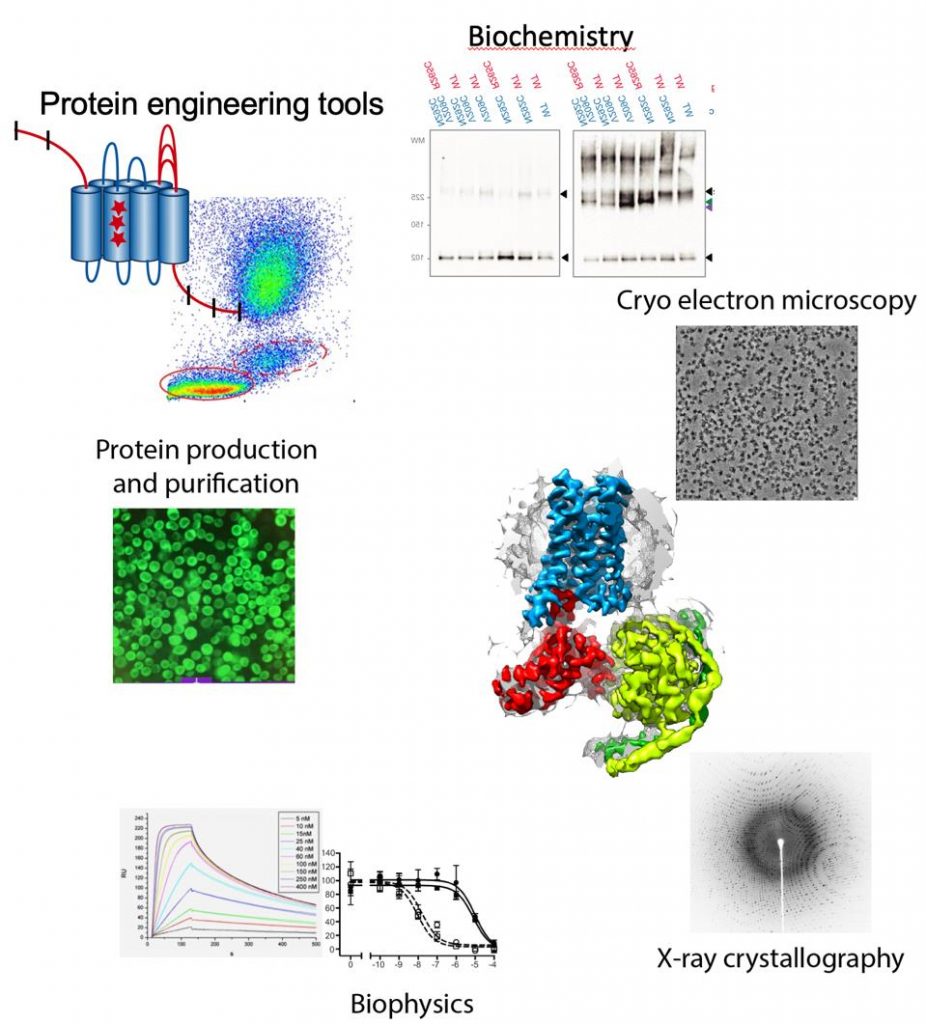
Las proteínas de membrana representan el 30% del genoma humano, son responsables de la comunicación intercelular y tienen una gran relevancia médica, con el 60% de los fármacos usando una proteína de membrana como diana. Las proteínas de membrana juegan un papel especialmente importante en el cerebro, donde la comunicación intercelular es la base de su función y da lugar a la formación de memoria y la consciencia. El objetivo del grupo es entender como los receptores neuronales funcionan, diseccionando como detectan estímulos externos y los transducen al interior celular. Tenemos especial interés en entender la integración de señales a través de complejos de receptores, que vuelven muy compleja la interpretación de señales, pero además son una nueva ruta para desarrolar fármacos mas específicos. Para llevar a cabo la investigación usamos un enfoque integrado de biología estructural (crio-microscopía electrónica y cristalografía de rayos X), biofísica y ensayos bioquímicos. La crio-microscopía electrónica de alta resolución juega un papel importante es nuestros estudios ya que podemos determinar estructuras de proteínas de membrana que antes eran inviables por cristalografía de rayos X.
 Herramientas de ingeniería de proteínas
Herramientas de ingeniería de proteínas
Las proteínas de membrana son intrínsecamente inestables fuera del entorno de la membrana. Para solucionar este problema desarrollamos nuestras herramientas de ingeniería de proteínas que tienen como fín estabilizar el plegamiento y maximizar la expresión de proteínas. Recientemente hemos desarrollado un sistema de clonaje que simplifica todos los protocolos de modificación de genes en plásmidos (García-Nafría, Sci. Rep, 2016 and Watson, JBC, 2019). Esto permite crear, de manera simple y rápida, crear librerías de cDNA de proteínas que se usan a posteriori para encontrar modificaciones estabilizantes. Este sistema se puede usar en cualquier laboratorio biomédico y con cualquier tipo de proteína expresada.
 Metodologías utilizadas
Metodologías utilizadas
- Clonación de genes y técnicas de DNA recombinante.
- Ingeniería de proteínas.
- Producción de proteínas en bacteria, células de insecto y células de mamífero.
- Purificación de proteínas.
- Resonancia de plasmón superficial (Biacore).
- Microscale thermophoresis.
- Cristalografía y difracción de rayos X de macromoléculas.
- Crio-microscopía electrónica de partícula única de alta resolución.
- Ensayos bioquímicos y funcionales en células.
Relevant publications
1. New insights into GPCR coupling and dimerisation from cryo-EM structures. Gusach A, García-Nafría J, Tate CG. Curr Opin Struct Biol. 2023 Mar 22;80:102574.
2. Molecular Cloning Using In Vivo DNA Assembly. Arroyo-Urea S, Watson JF, García-Nafría J. Methods Mol Biol. 2023;2633:33-44.
3. Molecular determinants of β-arrestin coupling to formoterol-bound β1-adrenoceptor.Lee Y., Warne T., Nehme R., Pandey S., Chaturvedi M., Dwivedi-Agnihotri H., Edwards P., García-Nafría J., Leslie A., Shukla AK., Tate CG. Nature. 2020 Jun 17. doi: 10.1038/s41586-020-2419-1.
4.Cryo-Electron Microscopy: Moving Beyond X-ray Crystal Structures for Drug receptors and Drug development. García-Nafría J.* and Tate CG*. Annual Reviews in Pharmacology and Toxicology. 2020. Jan 6;60:51-71.
5. In vivo DNA assembly using common laboratory bacteria: a re-emerging tool to simplify molecular cloning.Jake F. Watson and García-Nafría J. Journal of Biological Chemistry. 2019 Oct 18;294(42):15271-15281.
6. Architecture of the heteromeric GluA1/2 AMPA receptor in complex with the auxiliary subunit TARP γ8. Herguedas B, Watson JF, Ho H, Cais O, García-Nafría J, Greger IH. Science. 2019 Mar 14. pii: eaav9011
7. Cryo-EM structure of the serotonin 5-HT1B receptor coupled to heterotrimeric Go. García-Nafría J., Nehme R., Edwards P., Tate CG. Nature.2018, 558 (7711). 620-62.
8. Cryo-EM structure of the adenosine A2Areceptor coupled to an engineered heterotrimeric G protein. García-Nafría J., Lee Y., Bai X., Carpenter B. & Tate CG. Elife.2018, May 4;7. pii: e35946.
9. Structure and organization of heteromeric AMPA-type glutamate receptors. Herguedas B*, García-Nafria J*, Cais O, Fernandez-Leiro R, Ho H, Krieger J, Greger IH. Science.2016, Apr 29;352(6285):aad3873.
10. IVA cloning: A single-tube universal cloning system exploiting bacterial In Vivo Assembly. García-Nafria J*, Watson JF, Greger IH. Scientific Reports.2016, Jun 6;6:27459. (*corresponding author).
Main research projects
1. Ministerio de Ciencia e Innovación. In vitro de-orphanization of G protein-coupled receptors. MICINN. CNS2022-135877. 2023-2025.
2. Ministerio de Ciencia, Innovacion y Universidades. New modulation mechanisms of GPCRs (DopDrug): a focus on the dopamine receptors. MICINN. PID2020-113359GA-I00. 2021-2024.
3. Sosei Heptares Therapeutics. Optimization of sample production geared towards cryo-EM structure pipeline: application to orphan GPCRs. 2023-2026. Heptares Therapeutics limited.
4. Dotación adicional Ramón y Cajal. RYC2018-025731-I. 2020-2025.
Colaboradores
Paula Morales, Nadine Jagerovic (IQM, CSIC, Spain)
Amy H. Newman, Alessandro Bonifazi (NIDA, NIH, USA)
Maria José Sanchez Barrena (CSIC, Madrid, Spain).
Nunilo Cremades Casasín (BIFI, Zaragoza, Spain).
CONTACTO
Email: jgarcianafria@unizar.es
Pagina web del grupo: https://sites.google.com/view/signal-transduction-lab/
Modulación Enzimática & Mecanismos de Reacción
Responsable de la Línea de Investigación:
Pedro Merino Filella
Investigadores:
Manuel Pedrón (contratado pre-doctoral Gobierno de Aragón)
Sara Orta (contratado pre-doctoral Gobierno de Aragón)
Sandra Pereira (contratado pre-doctoral FPI)
Ignacio Sanz (contratado pre-doctoral FPI)
Colaboradores:
Tomas Tejero Lopez (ISQCH)
Iñaki Delso (ISQCH)
RESUMEN
SUBLINEA 1: Modulación y mecanismos de reacción enzimáticos
Nuestro grupo centra su actividad en conseguir comprender los procesos biológicos relacionados con la salud. El grupo posee una dilatada experiencia en síntesis orgánica asimétrica y cuenta con laboratorios bien equipados en la Facultad de Ciencias. Dentro del campo de la Química Biológica el grupo está interesado en el diseño y síntesis de pequeñas moléculas -principalmente compuestos nitrogenados y glicomiméticos- capaces de actuar como moduladores y/o inhibidores de enzimas diana asociados con procesos biológicos específicos. Entre otros enzimas, glicosiltransferasas y transglicosilasas son los principales objetivos. El desarrollo de estas actividades se lleva a cabo combinando una serie de técnicas multidisciplinares que incluyen metodologías sintéticas desarrolladas en nuestros laboratorios utilizando métodos de catálisis asimétrica orgánica y metálica, técnicas de biocomputación (docking y dinámica molecular) y técnicas espectroscópicas avanzadas como STD-NMR.
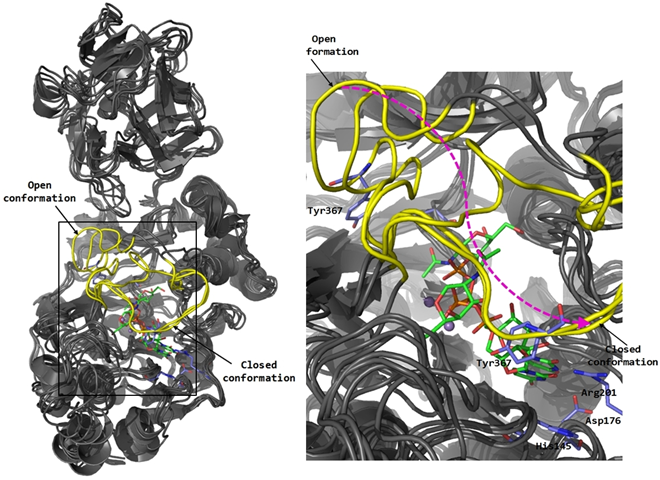 Figura 1. Glicosiltransferasa GalNAc-T2: Estudio de dinámica molecular e interacciones con un ligando diseñado en el grupo de investigación.
Figura 1. Glicosiltransferasa GalNAc-T2: Estudio de dinámica molecular e interacciones con un ligando diseñado en el grupo de investigación.
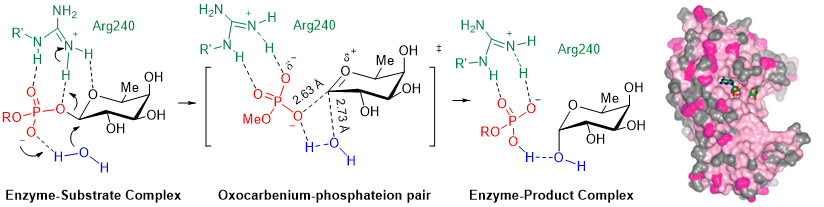 Figura 2. Glicosiltransferasa POFUT1: Mecanismo catalítico de tipo SN1 con formación de un par iónico en el estado de transición
Figura 2. Glicosiltransferasa POFUT1: Mecanismo catalítico de tipo SN1 con formación de un par iónico en el estado de transición
SUBLINEA 2: Mecanismos de reacciones orgánicas.
El grupo está también interesado en estudiar mecanismos de reacción utilizando mecánica cuántica y aproximaciones topológicas modernas como análisis ELF (función de localización electrónica) y NCI (interacciones no covalentes)
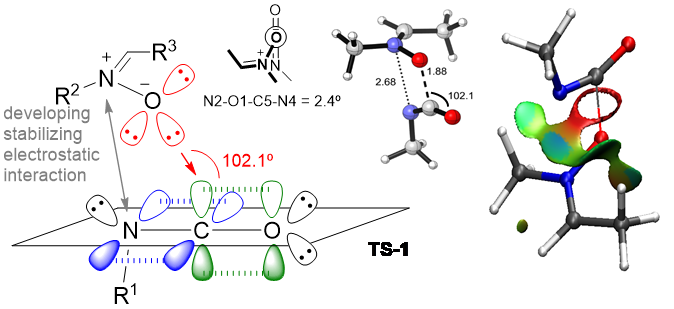 Figura 3. Estudios QM y topológicos de mecanismos de reacción
Figura 3. Estudios QM y topológicos de mecanismos de reacción
Los estudios incluyen la elucidación de reacciones catalíticas, tanto catalizadas por metales como organocatalítcas. En particular, el grupo se dedica a estudiar el rol de ácidos fosfóricos quirales en reacciones organocatalíticas en las que se forman carbocationes transientes.
PUBLICACIONES RELEVANTES
- Computational evidence of Glycosy Cations. Merino, P.; Delso, I.; Pereira, S.; Pedron, M.; Orta, S.; Tejero, T. Org. Biomol. Chem. 2021. doi: 10.1039/D0OB02373F
- Enantio- and Diastereoselective Nucleophilic Addition of N-tert-Butyl Hydrazones to Isoquinolinium Ions through Anion-Binding Catalysis. Matador, E.; Iglesias-Sigüenza, J.; Monge, D.; Merino, P.; Fernández, R.; Lassaletta, J. M. Angew. Chem. Int. Ed. 2021,133, 5096-5101. doi: 10.1002/anie.202012861
- Anomeric beta-triflate characterization enables the monitorization of glycosylation dynamics and suggests a non-canonical reinterpretation of the mechanism. Santana, A. G.; Montalvillo-Jiménez, L.; Díaz-Casado, L.; Corzana, F.; Jiménez-Osés, G.; Merino, P.; Cañada, F. J.; Jiménez-Barbero, J.; Gómez, A. M.; Asensio, J. L. J. Am. Chem. Soc. 2020, 142, 12501-12514. doi: 10.1021/jacs.0c05525
- Enantioselective Synthesis of Tropanes through Brønsted Acid-Catalyzed Pseudotransannular Desymmetrization. Rodriguez, S.; Uria, U.; Reyes, E.; Carrillo, L.; Tejero, T.; Merino, P.; Vicario, J. L. Angew. Chem. Int. Ed. 2020, 132, 6846-6850. doi: 10.1002/anie.202000650
- Dissecting the Structural and Chemical Determinants of the “Open-to-Closed” Motion in the Mannosyltransferase PimA from Mycobacteria. Unzueta, A. R.; Ghirardello, M.; Urresti, S.; Delso, I.; Giganti, D.; Anso-Miqueleiz, I.; Trastoy, B.; Comino, N.; Tersa, M.; D’Angelo, C.; Cifuente, J. O.; Marina, A.; Durana, A.; Chenal, A.; Svergun, D. I.; Alzari, P. M.; Albesa-Jové, D.; Merino, P.; Guerin, M. E. Biochemistry 2020, 59, 2934-2945. doi: 10.1021/acs.biochem.0c00376
- Enantioselective Synthesis, DFT Calculations and Preliminary Antineoplastic Activity of Dibenzo 1-Azaspiro[4.5]decanes on Drug Resistant Leukemias. Mendes, J. A.; Merino, P.; Soler, T.; Salustiano, E. J.; Costa, P. R. R.; Yus, M.; Foubelo, F.; Buarque, C. D. J. Org. Chem. 2019, 84, 2219-2233. doi: 10.1021/acs.joc.8b03203
- Sequential Metal-free Thermal 1,3-Dipolar Cycloaddition of Unactivated Azomethine Ylides. Selva, V.; Selva, E.; Merino, P.; Nájera, C.; Sansano, J. M. Org. Lett. 2018, 20, 3522-3526. doi: 10.1021/acs.orglett.8b01292
- Catalytic Enantioselective Cloke-Wilson Rearrangement. Ortega, A.; Manzano, R.; Uria, U.; Carrillo, L.; Reyes, E.; Tejero, T.; Merino, P.; Vicario, J. L. Angew. Chem. Int. Ed. 2018, 57, 8225-8229. doi: 10.1002/anie.201804614
- UDP-GlcNAc Analogs as Inhibitors of O-GlcNAc Transferase (OGT): Spectroscopic, Computational and Biological Studies. Ghirardello, M.; Perrone, D.; Chinaglia, N.; Sádaba, D.; Delso, I.; Tejero, T.; Marchesi, E.; Fogagnolo, M.; Rafie, K.; Aalten, D. M. F. v.; Merino, P. Chem. Eur. J. 2018, 24, 7264-7272. doi: 10.1002/chem.201801083
- Inhibitors against Fungal Cell Wall-remodelling Enzymes. Delso, I.; Valero-Gonzalez, J.; Gomollón-Bel, F.; Castro-López, J.; Fang, W.; Navratilova, I.; Aalten, D. M. F. v.; Tejero, T.; Merino, P.; Hurtado-Guerrero, R. ChemMedChem 2018, 13, 128-132. doi: 10.1002/cmdc.201700720
- (+)-Methyl (1R,2S)-2-{[4-(4-chlorophenyl)-4-hydroxypiperidin-1-yl]methyl}-1-phenylcyclopropanecarboxylate [(+)-MR200] Derivatives as Potent and Selective Sigma Receptor Ligands: Stereochemistry and Pharmacological Properties. Amata, E.; Rescifina, A.; Prezzavento, O.; Arena, E.; Dichiara, M.; Pittalà, V.; Montilla-García, Á.; Punzo, F.; Merino, P.; Cobos, E. J.; Marrazzo, A. J. Med. Chem. 2018, 61, 372-384. doi: 10.1021/acs.jmedchem.7b01584
- Regioselectivity Change in the Organocatalytic Enantioselective (3+2) Cycloaddition with Nitrones Through Cooperative H-Bonding Catalysis/Iminium Activation. Prieto, L.; Juste-Navarro, V.; Uria, U.; Delso, I.; Reyes, E.; Tejero, T.; Carrillo, L.; Merino, P.; Vicario, J. L. Chem. Eur. J. 2017, 23, 2764-2768. doi: 10.1002@chem.201605350
- Racemic Hemiacetals as Oxygen-Centered Pronucleophiles Triggering Cascade 1,4-Addition/Michael Reaction through Dynamic Kinetic Resolution under Iminium catalysis. Reaction Development and Mechanistic Insights. Orue, A.; Uria, U.; Roca-López, D.; Delso, I.; Reyes, E.; Carrillo, L.; Merino, P.; Vicario, J. L. Chem. Sci. 2017, 8, 2904-2913. doi: 10.1039/C7SC00009J
- The small molecule luteolin inhibits N-acetyl--galactosaminyltransferases and reduces mucin-type O-glycosylation of amyloid precursor protein Liu, F.; X, K.; Xu, Z.; Rivas, M. d. l.; Li, X.; Lu, J.; Delso, I.; Merino, P.; Hurtado-Guerrero, R.; Zhang, Y. Journal of Biological Chemistry 2017, 292, 21304-21319. doi: 10.1074/jbc.M117.814202
PRINCIPALES PROYECTOS DE INVESTIGACIÓN
1.- Diseño racional y síntesis estereoselectiva de glicomiméticos. MINECO, 2014-2016 CTQ2013-44367-C2-1-P. 151.200 EURO. PI: Pedro Merino
2.- Diseño racional de glicomiméticos inhibidores de glicosiltransferasas. MINECO, 2017-2019 CTQ2016-76155-R. 172.800 EURO. PI: Pedro Merino
3. Estudios mecanísticos de reacciones de glicosilación y su aplicación al diseño de inhibidores de glicosiltransferasas. Ministerio de Ciencia, Innovacion y Universidades. PID2019-104090RB-100. 2020-2023. 163.350 EUROS PI: Pedro Merino Filella
COLABORADORES
Dr. Loredana Maiuolo. Università della Calabria. Cosenza. Italy
Prof. Jose Luis Vicario. Universidad del Pais Vasco. Bilbao. Spain
Prof. Jose M. Lassaaletta. IIQ-CSIC. Sevilla. Spain.
Dr. Francesca Cardona. Universitá di Firenze. Italy
Prof. Dan van Aalten. University of Dundee. United Kingdom.
Prof. Sonsoles Martin-Santamaría. CIB-CSIC. Madrid. Spain.
Prof. Juan Luis Asensio. IQO-CSIC, Madrid. Spain
Prof. Eric Oldfield. University of Illinois, USA
Dr. Francisco Corzana. Universidad de La Rioja. Spain
Dr. Marcelo Guerin. CIC-Biogune. Bilbao. Spain
Prof. Pedro Gois. Universidade de Coimbra. Portugal.
Dr. Ramon Hurtado. Universidad de Zaragoza. BIFI. Spain
CONTACTO (opcional email, web personal o grupo, etc)
pmerino@unizar.es
bioorganica@unizar.es